Conocimientos previos:
- ADN
- ARN
- Edición del ARN
- Transcripción y Traducción
- Expresión génica
- Organelos
- Eucariotas y Procariotas
Resumen
Un gen es una región de ADN que controla una característica hereditaria, que por lo general corresponde a una molécula de una proteína o una secuencia de ARN. Existen genes que codifican ARNm monocistrónicos y policistrónicos que son los que expresan una o varias proteínas respectivamente. Para que un gen pueda producir una proteína es necesario producir un ARNm maduro eliminando los intrones y traducirlo mediante el uso del código genético. El estudio de los genes y su expresión ha llevado a los biólogos a poder manipularlo y crear herramientas que permiten su edición. Estas novedosas herramientas tienen implicaciones en los campos de la medicina y la industria, pero su aplicación en el humano ha puesto en un debate bioético a la comunidad científica. Esta tecnología de reciente advenimiento permitirán revolucionar el conocimiento de los genes para su aplicación y el mejoramiento de la calidad de vida del ser humano.
Introducción
Un gen es una región de ADN y controla una característica hereditaria, que por lo general corresponde a una molécula de una proteína o un ARN. Los genes están vinculados con el desarrollo o funcionamiento de una función fisiológica de la célula (Alberts et al., 2014; Lewin, 2004).
La información genética de las células eucarióticas se almacena en el núcleo, pero una pequeña cantidad permanece dentro de las mitocondrias y en los cloroplastos en las células de plantas y algas. Esto es porque estas células tienen un origen híbrido según la teoría de endosimbiosis; un ancestro eucariota anaeróbico y una bacteria que se internalizó como su simbionte para formar estos organelos (Sagan, 1967).
El gen es considerado como la unidad de almacenamiento de información genética y de herencia, ya que, transmite esa información de padres a hijos. Cada organismo de reproducción sexual cuenta con dos copias de material genético, una proveniente del padre y otra de la madre; esto permite tener dos versiones distintas de un mismo gen. Los genes pueden tener versiones diferentes, con pequeñas variaciones en su secuencia a esto se le denomina alelos. Los alelos pueden ser dominantes o recesivos. Cuando una sola copia de un alelo hace que se manifieste un rasgo o un fenotipo, el alelo se denomina dominante. Cuando son precisas dos copias del alelo, para que se manifieste su efecto, el alelo se denomina recesivo (Alberts et al., 2014).
Los genes se disponen a lo largo de los cromosomas y ocupan una posición específica llamada locus. Los genes que no codifican a proteínas pueden producir un ARN funcional, como el ARN de transferencia (ARNt) o el ARN ribosomal (ARNr). Sin embargo, para generar una proteína se requiere una molécula de ARNm que posteriormente se traducirá en un organelo llamado ribosoma, donde se generarán las proteínas. Muchos genes se encuentran constituidos por regiones codificantes llamadas exones y están flanqueadas por regiones no codificantes llamadas intrones. Estas secuencias pueden ser eliminadas en un proceso llamado “splicing” del ARN. En células bacterianas este procesamiento no ocurre, pues sus genes carecen de intrones. Los nucleótidos presentes en la secuencia del ARNm provenientes del gen determinara la secuencia de aminoácidos de una proteína por medio del código genético (Alberts et al., 2014).
Código genético
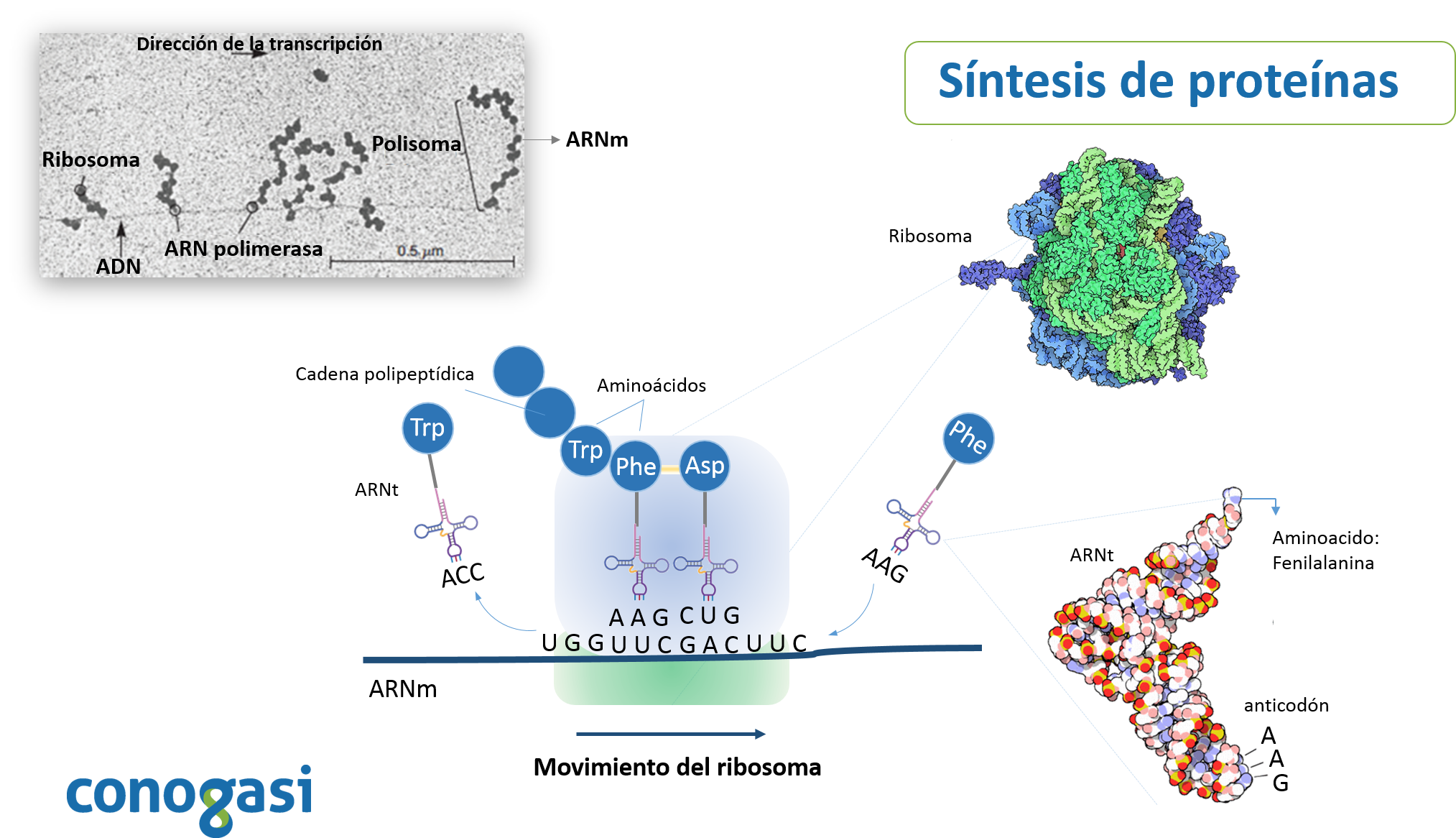
El código genético es el conjunto de reglas por las cuales la información codificada en el ADN se traduce a proteínas. Este proceso de desciframiento ocurre dentro de los ribosomas donde se lleva a cabo la traducción; los aminoácidos son llevados por las moléculas de ARN de transferencia (ARNt) y se unen en un orden que es especificado por el ARNm. El ARNt lee los ARNm de tres en tres nucleótidos y coloca un aminoácido para ir formando una proteína (figura 1) (Alberts et al., 2014).
Código Genético
El código genético en general es muy similar entre todos los organismos (tabla 1), y fueron necesarios diversos estudios para su dilucidación. Uno de ellos fue el experimento de Crick y Brenner sugiriendo que los codones consisten en 3 bases nitrogenadas esto lo demostró con el cistron B de la región rII del bacteriófago T4, el cual ataca a las cepas de E. coli. La región rII consiste en dos genes adyacentes o cistrones llamados A y B. Los fagos silvestres crecen en E. coli B (llamado B) y en E. coli K12 λ (llamado K) pero el fago mutante en r ha perdido la función en cada gen y no crece en K. Crick y Brenner trabajaron con un fago mutante (FCO) en el segmento B1 del cistrón B, que fue producido por la acción de la proflavina (La proflavina actúa como agente mutágeno insertando o deletando una o varias bases). Los bacteriofagos T4 mutantes producidos por Crick y Brenner no podían producir la proteína funcional rII B debido a la inserción o deleción de nucleótidos causando un desplazamiento de marco de lectura abierto del gen. Sin embargo, podían revertir la mutación y recuperar su fenotipo insertando o eliminando de nuevo nucleótidos con la proflavina. Con estos experimentos concluyen en su investigación y junto con otras aportaciones como las de Nirenberg y Matthaei, proponen que el código genético podría utilizar un codón de tres bases de ADN el cual corresponde a un aminoácido (F. H. Crick, Barnett, Brenner, & Watts-Tobin, 1961).
Tabla 1. Código genético estándar.
| Aminoácido | Codón | Aminoácido | Codón |
| Ala (A) | GCU, GCC, GCA, GCG | Lys (K) | AAA, AAG |
| Arg (R) | CGU, CGC, CGA, CGG, AGA, AGG | Met (M) | AUG |
| Asn (N) | AAU, AAC | Phe (F) | UUU, UUC |
| Asp (D) | GAU, GAC | Pro (P) | CCU, CCC, CCA, CCG |
| Cys (C) | UGU, UGC | Sec (U) | UGA |
| Gln (Q) | CAA, CAG | Ser (S) | UCU, UCC, UCA, UCG, AGU, AGC |
| Glu (E) | GAA, GAG | Thr (T) | ACU, ACC, ACA, ACG |
| Gly (G) | GGU, GGC, GGA, GGG | Trp (W) | UGG |
| His (H) | CAU, CAC | Tyr (Y) | UAU, UAC |
| Ile (I) | AUU, AUC, AUA | Val (V) | GUU, GUC, GUA, GUG |
| Leu (L) | UUA, UUG, CUU, CUC, CUA, CUG | ||
| Inicio | AUG | Paro | UAG, UGA, UAA |
| Codones de inicio alternativos en Eucariotes | Referencia | ||
| Candida albicans (hongos) | GUG proteína P | (Abramczyk, Tchórzewski, & Grankowski, 2003) | |
| Mamiferos | GUG proteína NAT1 | (Takahashi et al., 2005) | |
| Codones de inicio alternativos en Mitocondrias de Eucariotes | Referencia | ||
| Bos taurus: bovino | AUA | (Elzanowski, Andrzej (Anjay), 2010) | |
| Homo sapiens: Humano | AUA, AUU, AUG | (Anderson et al., 1981) | |
| Mus musculus: Ratón | AUA, AUU, AUC | (Bibb, Van Etten, Wright, Walberg, & Clayton, 1981) | |
| Gallus gallus: gallo | AUA, AUU, AUC, GUG | (Desjardins & Morais, 1991) | |
Posteriormente, Marshall Nirenberg y Heinrich Matthaei publican sus resultados sobre la decodificación del código genética en 1961. Utilizando un sistema sin células, el cual podía traducir una secuencia de ARN. Utilizaron una secuencia de ARN de poliuracilo (UUUUU(n)) y descubrieron que el polipéptido que se sintetizó contenía fenilalanina. Demostraron además que la síntesis de proteínas es dependiente de energía y se inhibe por la presencia de RNasas, puromicina y cloranfenicol dejando además evidencias de la existencia de un ARNm (Nirenberg & Matthaei, 1961). Posteriormente, Nirenberg y Philip Leder determinaron que el código genético estándar está formado por codones los cuales contienen 3 bases nucleotídicas. En estos experimentos, se hicieron pasar varias combinaciones de secuencias cortas de ARN a través de un papel filtro que contenía los ribosomas de E. coli y una combinación de aminoácidos marcados con carbono 14 unidos a un ARN (C14-AA-ARNs) (Figura 2). La mayoría de los trinucleótidos estimuló la unión al ribosoma con un sólo C14-AA-ARNs. Leder y Nirenberg fueron capaces de determinar las secuencias de 54 de los 64 codones en sus experimentos. Subsecuentemente Har Gobind Khorana, ideó métodos bioquímicos precisos e intrincados para producir cadenas de nucleótidos para luego sintetizar un ARN sintético usando una polimerasa para después traducirlos en cadenas de aminoácidos (Figura 3).
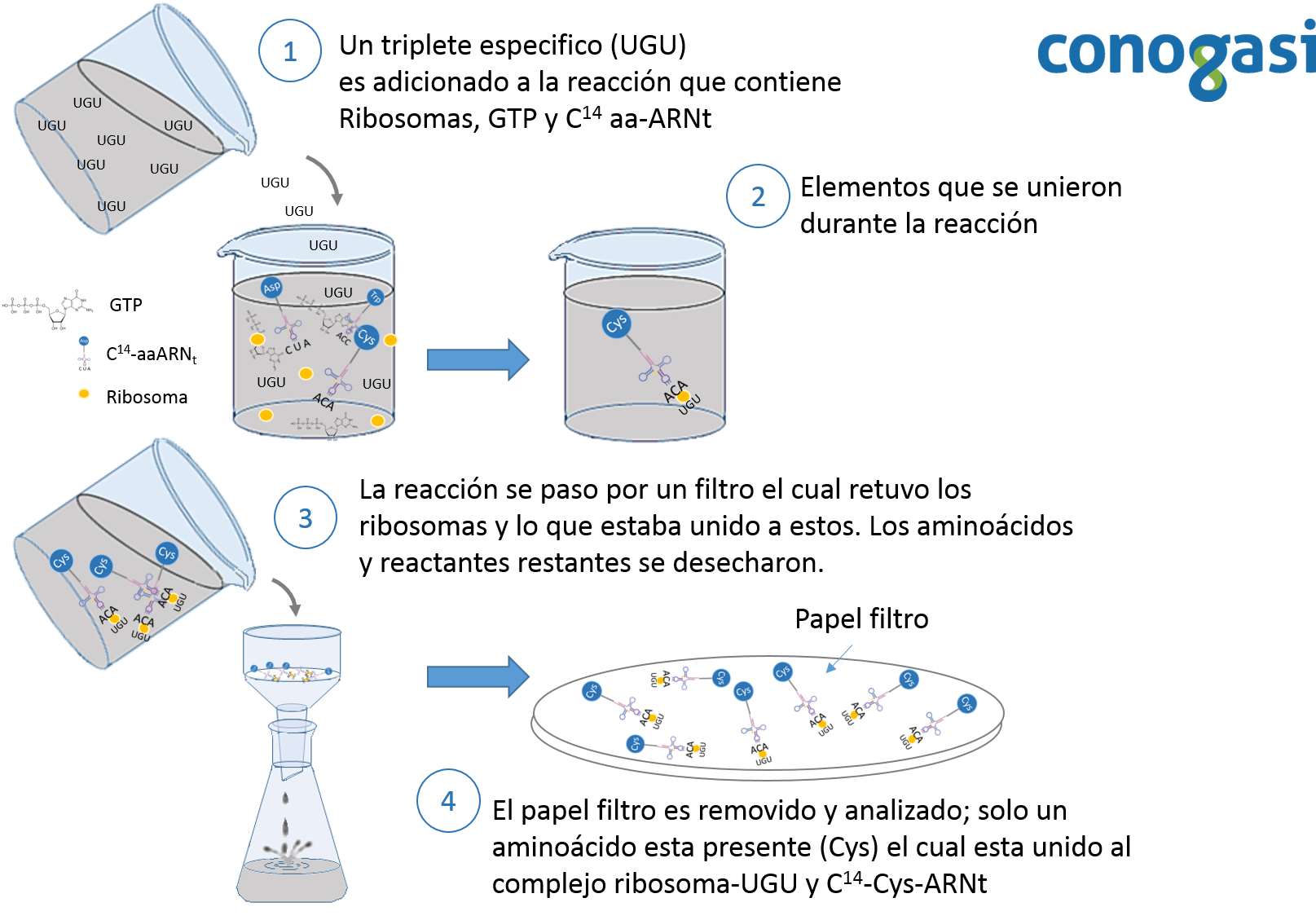
Estos experimentos lograron interpretar parte del código genético. Ese mismo año, Severo Ochoa demostró por otra parte en una serie de artículos en la revista PNAS (Proceedings of National Academy of Science) que sintetizando diferentes secuencias del ARN y traducirlas en sistemas libres de células obtuvo resultados similares como: cadenas de AAA, daba como resultado polilisina; cadenas de 2A1U, asparagina, isoleucina, lisina; cadenas de 1U2A, isoleucina, leucina, tirosina; y UUU, fenilalanina (Lengyel, Speyer, & Ochoa, 1961). Clark y Marcker mostraron que la síntesis de proteínas inicia mediante el reconocimiento del codón AUG, mientras que este mismo codón en posiciones internas corresponde a metionina (Clark & Marcker, 1966). Sidney Brenner, Alan Garen y sus colaboradores informaron que los codones UAA, UAG y UGA corresponden a la terminación de la síntesis de proteínas (Brenner, Stresson, & Kaplan, 1965).
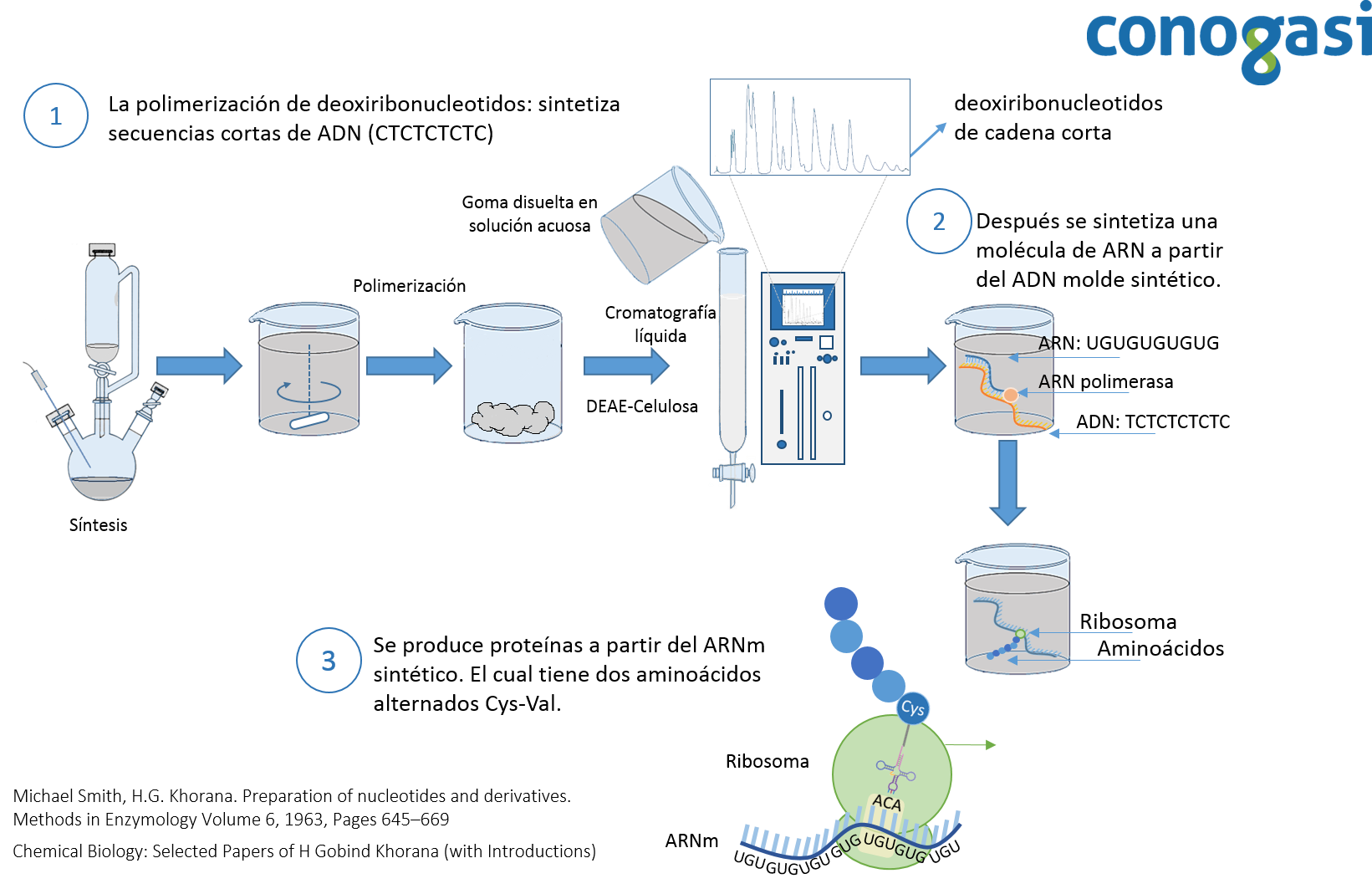
Por otra parte, Robert Holley de la Universidad de Cornell, descubrió un tipo especial de ácido nucleico llamado ARN de transferencia (ARNt). Holley fue capaz de secuenciar y determinar la estructura exacta del ARNt-alanina, la molécula adaptadora que facilita el proceso de traducción del ARN en proteína. Esta fue la primera vez que alguien había establecido la estructura química completa de una secuencia de ácidos nucleicos biológicamente activa (Holley et al., 1965). Finalmente, H.G. Khorana hizo otra importante aportación para la genética moderna siendo un hito en este campo, ya que produjo el primer gen sintético en 1970, y nueve años después demostró su funcionalidad en un organismo (Cech, 2011).
Origen, Estructura y Función de los Genes
La gran mayoría de los organismos usan esencialmente el mismo código genético, referido comúnmente como el código genético estándar. Esto se demostró purificado fracciones ARNt de bacterias, anfibios y mamíferos. Se comparó el código genético de E. coli con el del anfibio Xenopus leavis (rana africana) y con el de Cavia porcellus (cobayos), encontrando que el código es esencialmente universal (Marshall, Caskey, & Nirenberg, 1967). Aunque existen variaciones en algunos organismos se reconocen 64 codones para 20 aminoácidos y señales de inicio y paro con modificaciones en algunos organismos que han evolucionado el código y cambiado el código para algunos genes que codifican para proteínas (ver tabla 1) (Hinegardner & Engelberg, 1963). Durante la traducción de proteínas la fidelidad es muy alta durante la fase de elongación, ya que el ribosoma posee mecanismos especiales para lograrlo (Moore & Steitz, 2002). Aunque la traducción en eucariontes generalmente comienza con el codón AUG, en procariotas se permiten los codones GUG y UUG. En E. coli (gram negativos), AUG, GUG y UUG comienzan la traducción de sus proteínas en un 83, 14 y 3% respectivamente. Mientras que en B. subtilis (gram positivo), estos codones comienzan la traducción de proteínas en un 78, 9 y 13% (Rocha, Danchin, & Viari, 1999). Por otro lado, las arqueas muestran niveles similares de iniciación de UUG y GUG (Torarinsson, Klenk, & Garrett, 2005). Esto se debe en gran parte a un problema fundamental para el ribosoma para reconocer con precisión el codón de inicio. Sin embargo, la fidelidad en la iniciación de la traducción varía ampliamente entre los diferentes organismos.
Incluso una observación superficial de la tabla del código genético estándar muestra que la disposición de asignaciones de aminoácidos no es aleatoria (ver tabla 1) (Woese, 1965b). Generalmente, los codones que difieren por un solo nucleótido tienden a codificar para aminoácidos relacionados, es decir, aminoácidos que son físico-químicamente similares (aunque no existen criterios indiscutibles para definir la similitud fisicoquímica). La cuestión fundamental es cómo estas regularidades del código estándar entró en vigor desde su origen (Chechetkin, 2003). Por otro lado, se han planteado diversas hipótesis sobre el origen en común que tiene el código genético con todos los seres vivos. Los tres conceptos principales sobre el origen y evolución del código son la teoría estereoquímica (Gamow, 1954; Pelc, 1965; Woese, 1965a), la cual dice que las asignaciones de los codones son dictadas por una afinidad fisicoquímica entre los aminoácidos y los codones afines (anticodones). La teoría de la coevolución, que postula que la estructura del código coevolucionó con las vías de biosíntesis de aminoácidos; y la teoría de minimización de errores en las que la selección minimiza el efecto adverso de las mutaciones puntuales y los errores de traducción eran el principal factor de la evolución del código.
Estas teorías no son excluyentes y son además compatibles con la hipótesis del accidente congelado de F. Crick, el cual dice que el código es universal porque en la actualidad cualquier cambio sería letal. Esto se debe a que, en todos los organismos el código determina (mediante la lectura del ARNm) las secuencias de aminoácidos de las proteínas altamente evolucionadas, y cualquier cambio en estas sería altamente desventajoso a menos que estuviera acompañado de mutaciones simultáneas para corregir estos “errores” producidos por la alteración del código. Esto explicaría el hecho de que el código no cambia y se debe asumir que toda la vida evolucionó a partir de un solo organismo (más estrictamente, de una sola población estrechamente entrecruzada). En su forma extrema, la teoría implica que la asignación de los codones a los aminoácidos en este punto era enteramente una cuestión del “azar” (F. H. C. Crick, 1968). Dado el nivel de optimización relativamente modesto del código estándar, parece esencialmente correcto que la evolución del código implicó alguna combinación de accidente congelado con la selección para la minimización de errores, la estereoquímica y la coevolución.Estudios recientes de análisis de las trayectorias evolutivas del código muestra que es altamente resistente a errores en la lectura de la traducción y no pueden descartarse ningún concepto sobre el origen y evolución del código confirmando los conceptos anteriores. Ya que, gran parte de la evolución que condujo al código estándar, podría ser una combinación del accidente congelado, la estereoquímica y la coevolución. Pero aún es incierto cómo evolucionó y se originó el código genético (Novozhilov, Wolf, & Koonin, 2007). Además, si otros factores conocidos y/o aún desconocidos contribuyeron al origen y evolución del código genético, sigue siendo sujeto a investigación teórica, de modelado matemático y ha validación experimental.
Se ha propuesto en estudios teóricos que existe una relación entre la evolución de la secuencia de los genes, su expresión, y la producción de proteínas mal plegadas como un factor crucial en la evolución (D. Allan Drummond, Bloom, Adami, Wilke, & Arnold, 2005). Se asume que un mal plegamiento de proteínas podría impulsar la evolución de las secuencias de las proteínas y al propio código genético (D. Allan Drummond & Wilke, 2008). Actualmente existe demasiada evidencia de que el código estándar no es literalmente universal, sino que es propenso a modificaciones aunque, sin cambios en su organización básica (Koonin & Novozhilov, 2009). Desde el descubrimiento de la reasignación de codones en los genes mitocondriales humanos (Barrell, Bankier, & Drouin, 1979), se han reportado una variedad derivaciones del código genético estándar en otros organismos como bacterias, arqueas, eucariotas y especialmente en los genomas de sus organelos (Knight, Freeland, & Landweber, 2001; Sengupta, Yang, & Higgs, 2007). Los mayores cambios en el código genético se presentan en las mitocondrias de las células eucariotas con 25 modificaciones registradas en metazoarios, hongos, algas verdes, rojas y pardas, plantas verdes y protistas (Robin D. Knight, Landweber, & Yarus, 2001). En algunas bacterias y eucariotas hay alteraciones del código genético que implican, cambios sin asignación de codón, esto quiere decir que los hay codones con nuevas asignaciones para aminoácidos en sus genomas. Por ejemplo: en ciliados, los codones de paro (UAA y UAG) cambiaron a glutamina, o UAA a glutamato o UGA cambio a cisteína (Caron & Meyer, 1985)(Caron & Meyer, 1985; Harper & Jahn, 1989). Por otro lado en Bacillus subtilis los codones UGA se utilizan para terminar la traducción del ARNm (codón de paro) e insertar un triptófano este es el cambio más frecuente de reasignación del codón de paro UGA por el triptófano, esto implicaría que debe haber una predisposición a ciertos cambios que confieren una ventaja selectiva (Lovett, Ambulos, Mulbry, Noguchi, & Rogers, 1991; Santos, Cheesman, Costa, Moradas-Ferreira, & Tuite, 1999), y en Micrococcus luteus, Mycoplasma capricolum los codones AGA/AUA, CGG no están asignados, respectivamente (Kano, Ohama, Abe, & Osawa, 1993; Oba, Andachi, Muto, & Osawa, 1991).
La flexibilidad del código genético descrito anteriormente se hace aún más notorio por inserción del 21° aminoácido la selenocisteína en los sitios activos de las selenoproteínas en procariotas y eucariotas o por la inserción del 22° aminoácido la pirrolisina presente en la metiltransferasa monometilamina de la arquea Methanosarcina barkeri (Hao, 2011; Lee, Worland, Davis, Stadtman, & Hatfield, 1989). Estas ampliaciones a la reprogramación al código genético hacen resaltar la importancia de la expansión del código para generar innovación funcional y con ayuda de herramientas de última generación de biología sintética ha sido posible la creación de 49 nuevos aminoácidos no naturales que se han incorporado a organismos como E. coli, la levadura y células de mamíferos para producir nuevas proteínas de interés biotecnológico industrial y biomédico (Bacher, de Crecy-Lagard, & Schimmel, 2005; Bacher & Ellington, 2001; Jewett & Noireaux, 2016).
Operones
Los operones fueron descritos en 1960 por Jacob y Monod, como un grupo de genes bajo el control de una sola señal reguladora o promotor (Francois Jacob, David Perrin & Monod, 1960). Los genes son transcritos en una sola cadena de ARNm y se traducen juntos o se someten a trans–splicing para crear ARNm monocistrónicos que se traducirán por separado. El resultado en el caso de operones bacterianos, se demostró que los genes corregulados de un operón se expresan a partir de un ARN mensajero policistrónico único que se traduce en esa forma; sin embargo, la producción de ARNm policistrónico no forma parte de la definición de un operón y no es una propiedad de todo tipo de operones. Varios genes deben ser cotranscritos para definir un operón (Uzman, Lodish, Berk, Zipursky, & Baltimore, 2000)
Jacob y Monod pudieron distinguir entre dos tipos de secuencias en ADN en sus estudios: secuencias que codificanproductos que actúan de modo trans (usualmente proteínas) y las que actúan de modo cis (usualmente sitios en el ADN). La actividad de la expresión génica es regulada por interacciones específicas de los productos que actúan en modo trans con los que actúan de modo cis. La descripción de las secuencias en cis funciona exclusivamente como una secuencia in situ afectando solo el ADN el cual esta físicamente ligado (Lewin, 2004). Un ejemplo de una secuencia reguladora en cis es el operador en el operón lac. Esta secuencia de ADN está obligada por el represor lac que, a su vez, evita la transcripción de genes adyacentes en la misma molécula de ADN (figura 4). El operador lac es por lo tanto una secuencia que “actúa en cis” en la regulación de los genes cercanos. Los elementos cis son a menudo los sitios de unión de uno o más factores de transcripción. Este operador en si no codifica para ninguna proteína o ARN. Por el contrario, los elementos reguladores en trans son por lo general proteínas, que pueden modificar la expresión de un gen distante al gen que lo transcribió originalmente.
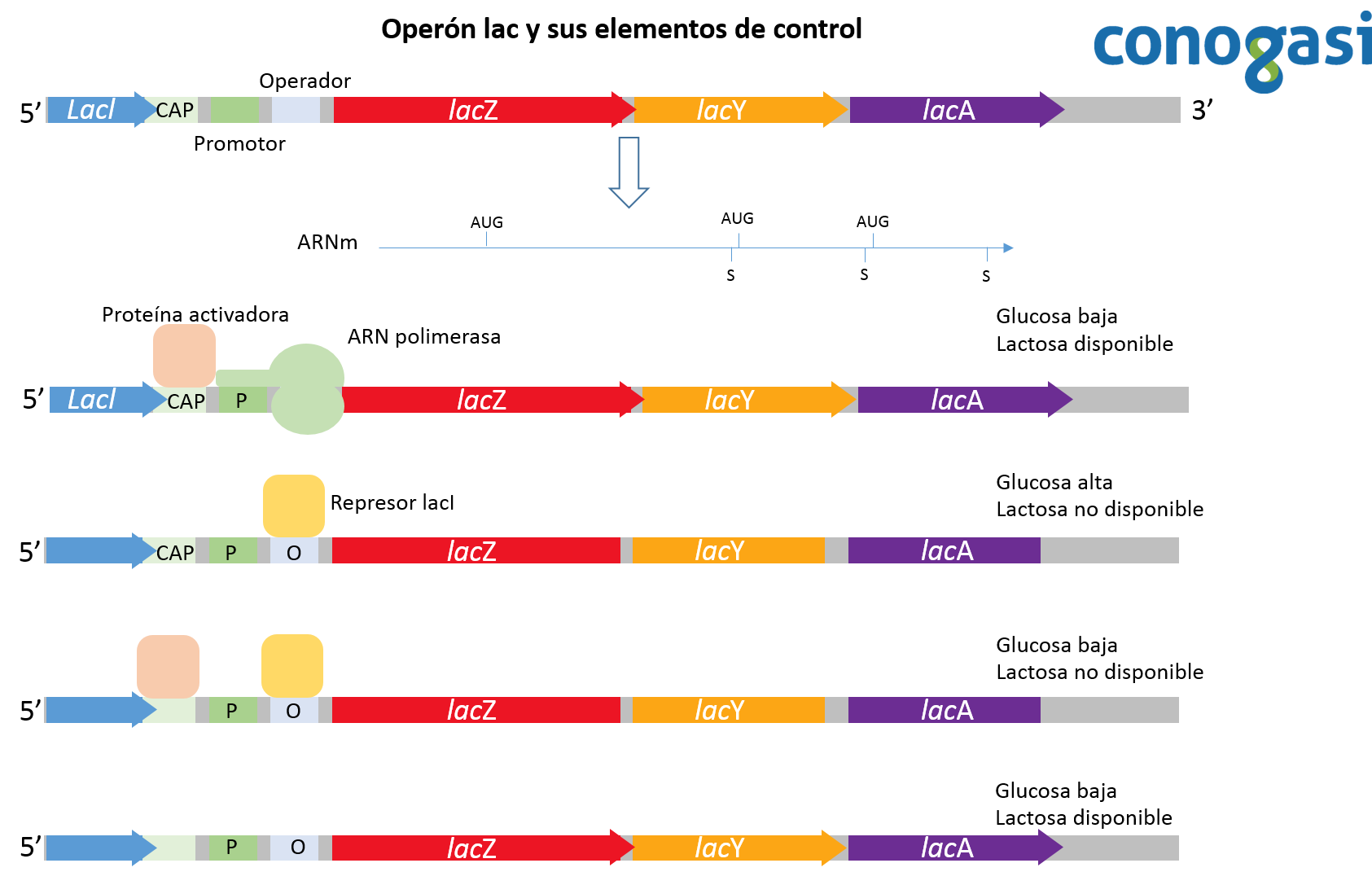
Un operón por lo tanto se define como una unidad genética funcional formada por un grupo de genes que ejercen una regulación en su expresión mediante la interacción de su secuencia sustrato y las proteínas que codifican sus mismos genes. Este complejo está formado por genes estructurales que codifican para la síntesis de proteínas (generalmente enzimas), que participan en vías del metabolismo y cuya expresión generalmente está regulada por otros tres factores de control:
- Promotor: sitio del operón con afinidad de unirse con la ARN polimerasa, este sitio controla el inicio de la transcripción. Un operón tiene un único promotor que controla toda su expresión, dando lugar a varias proteínas independientes.
- Gen regulador: dentro del operón uno de los genes puede codificar para un factor transcripcional que se unirá al promotor, regulando así la propia expresión del operón. A toda regulación de la expresión realizada desde dentro del gen u operón se le llama “regulación en cis”, pero puede haber también genes muy alejados del operón que codifican factores de transcripción para uno o varios genes u operones, y en este caso se hablaría de “regulación en trans”.
- Operador: zona de control que permite la activación/desactivación del promotor a modo de “interruptor genético” por medio de su interacción con un compuesto inductor. Tras su unión, por plegamientos tridimensionales interacciona con la zona del promotor, donde las proteínas reguladoras que se han unido contactan con la ARN polimerasa, aumentando o disminuyendo su afinidad por el promotor, y con ello dando lugar a la expresión/represión del resto de los genes estructurales.
ARN policistrónico
Anteriormente se pensaba que, la transcripción policistrónica era una característica específica de los procariontes (bacterias y arqueas) (figura 5), en donde muchos de los genes se agrupan en operones compuestos de dos a más de diez genes. Y, por el contrario, los genes de eucariotas se consideraban monocistrónicos, cada gen con su propio promotor en el extremo 5′ y un terminador de transcripción en el extremo 3′ (Figura 6). Sin embargo, se ha demostrado que no todos los genes eucariotas se transcriben como monocistrónicos. Se han descrito numerosos casos de transcripción policistrónica en eucariotas, desde protistas hasta cordados. Se han descrito tres tipos de operones: 1) presencia de transcritos dicistronicos; 2) el trans-splicing restringido usado dentro de los operones; 3) genes agrupados que son cotranscritos y tienen un proceso de trans-splicing (Blumenthal, 2004).
Dos genes relacionados y que se transcriben en un solo ARNm son GDF-1 y UOG-1, esta adaptación está conservada entre humanos y ratones. En algunos protozoarios como el Trypanosoma sp., toda la transcripción es policistrónica y todos los pre-ARNm son procesados mediante trans–splicing. En general, los genes en estas unidades de transcripción policistrónica carecen de intrones, por lo que el trans-splicing es el único splicing que sufren. Aunque no está claro si la mayoría de los conglomerados cotranscritos en los genomas de los tripanosomas están corregulados, existen ejemplos de genes funcionalmente relacionados que están cotranscritos (por ejemplo, cinco genes que codifican toda la ruta de novo de la biosíntesis de pirimidina) (Gao, Nara, Nakajima-Shimada, & Aoki, 1999).
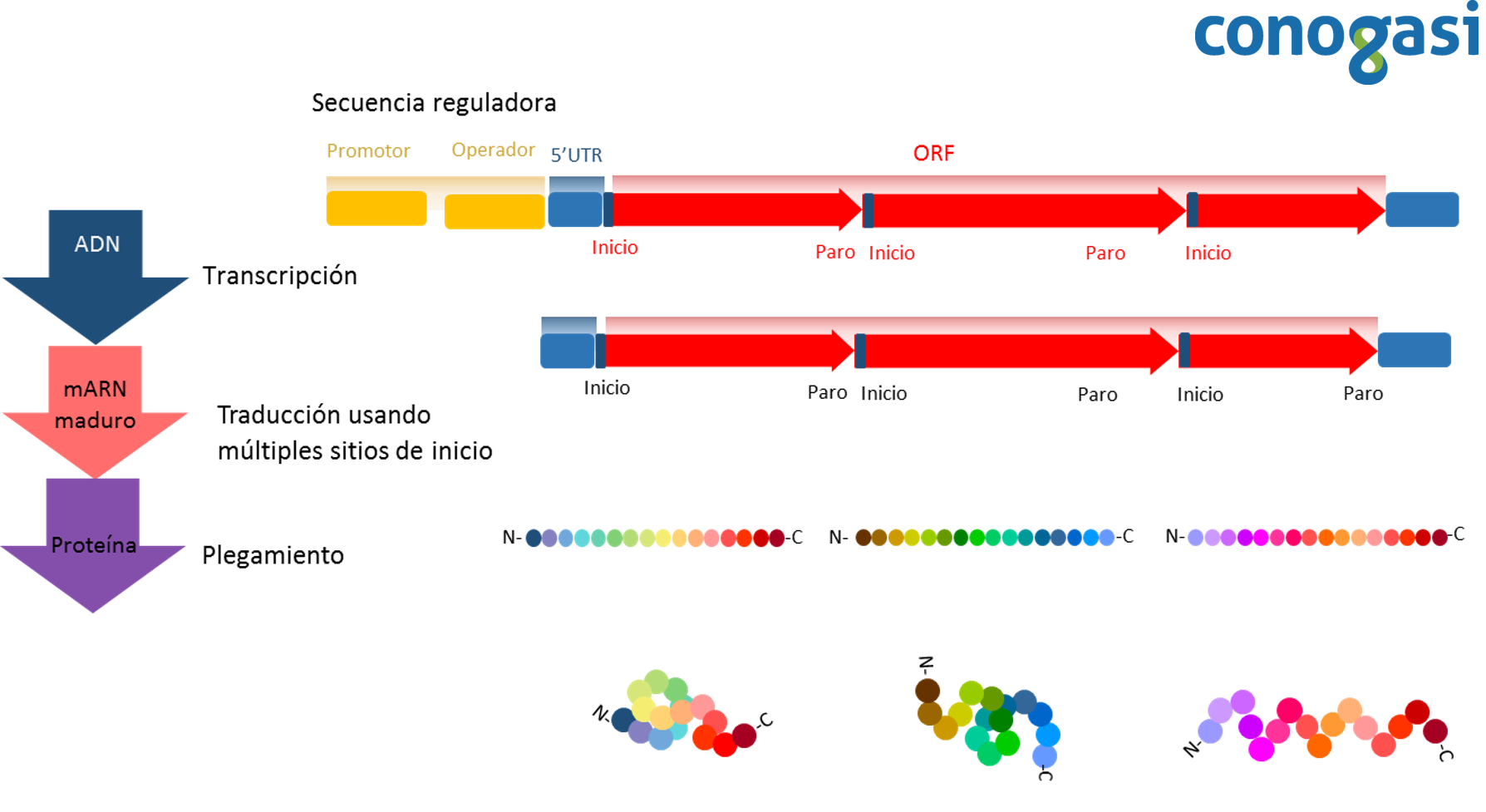
Por otro lado, los operones en los nematodos fueron descubiertos como una consecuencia de una fuerte correlación entre los genes con la misma orientación 5’ a 3’ existente en grupos cercanamente inusuales y la observación de que los genes río abajo fueran procesados por trans–splicing con un líder relativamente raro SL2 (Spieth, Brooke, Kuersten, Lea, & Blumenthal, 1993). Esta correlación resultó en la hipótesis de que los genes cotranscritos y que el SL2 era una forma de splicing especializado en la separación de los precursores de mRNAs policistrónicos. Trabajos posteriores demostraron que la hipótesis era correcta y que alrededor del 15 % de todos los genes de C. elegans se transcriben en agregados o grupos de transcritos policistrónicos que van de dos a ocho genes (Blumenthal et al., 2002; Zorio, Cheng, Blumenthal, & Spieth, 1994).
Estructura y función
La estructura de un gen está constituida por una secuencia de nucleótidos que componen varios elementos funcionales de los cuales generalmente solo una parte constituye la región que codifica una proteína. En bacterias y arqueas, muchos genes funcionalmente relacionados se organizan en operones para ser transcritos y traducidos simultáneamente. Los operones son poco conocidos en organismos eucariotas, pero se sabe que existen en protozoarios como los tripanosomas que son organismos unicelulares y nematodos o gusanos cilíndricos. En estos organismos se transcribe un ARN policistrónico, para después procesarse en ARNm maduros individuales. Se ha reportado también transcritos policistrónicos en algunos insectos. De forma similar a los operones de los procariotas, los péptidos codificados o las proteínas se traducen simultáneamente a partir de un solo ARNm policistrónico, proporcionando nuevos conocimientos sobre la evolución de los genes policistrónicos. Sor interesantemente, un tipo de los genes policistrónicos recién identificados codifica péptidos biológicamente importantes compuestos por tan sólo 11 aminoácidos (Pi, Lee, & Lo, 2009).
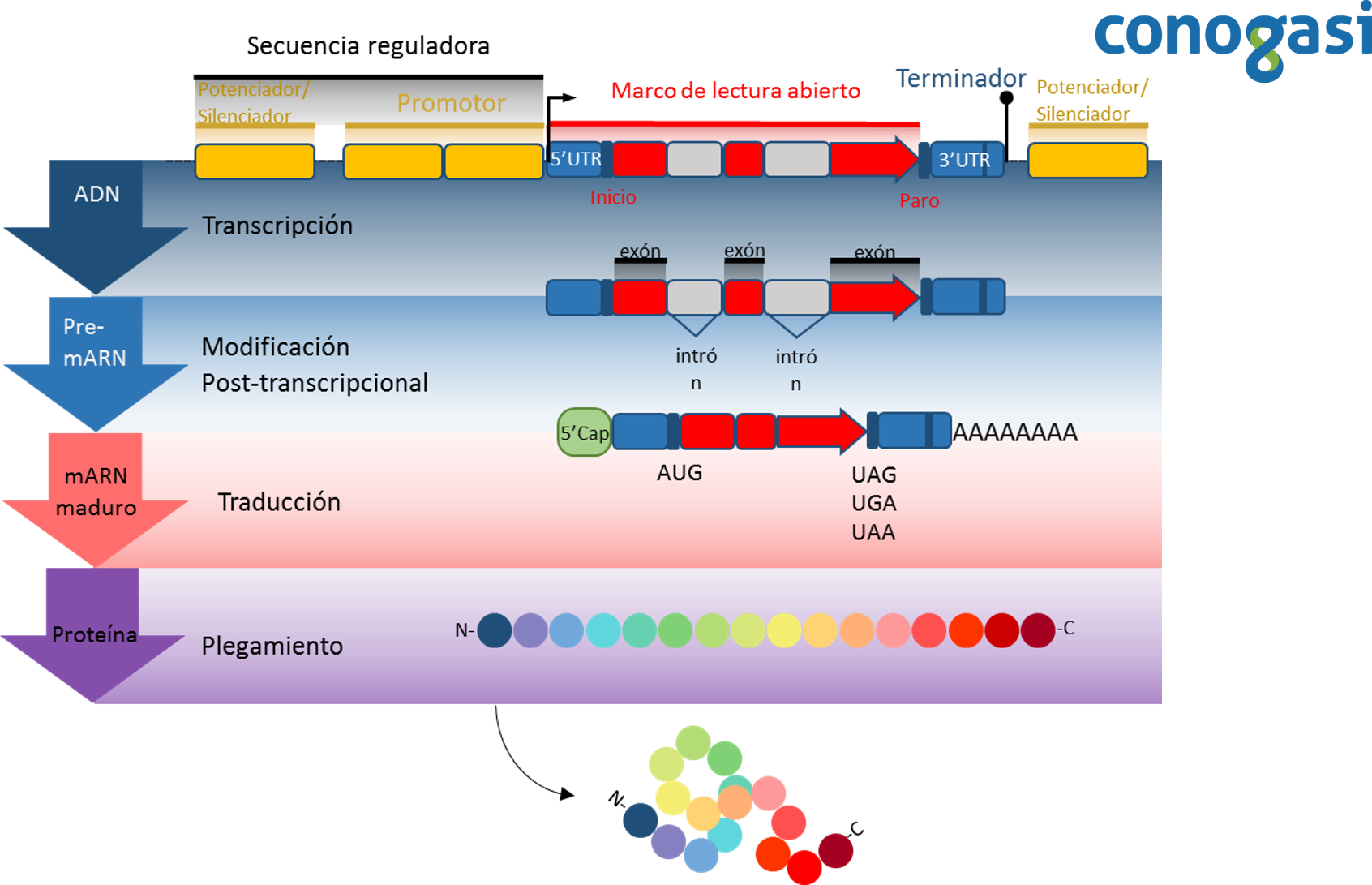
Las partes que constituyen a un gen son:
–Secuencia reguladora: Una secuencia reguladora es un segmento de ácidos nucleicos capaces de aumentar o disminuir la expresión de genes específicos dentro de un organismo.
–Potenciador/silenciador: Un potenciador es una región corta de ADN que influenciado por una proteína llamada “factor transcripcional” (Ft activadores) aumentan la probabilidad de que la transcripción de un gen se produzca. Por el contrario, un silenciador es una región de ADN que se unen a un Ft represor. Cuando un Ft represor se une a la región silenciador, el ARN polimerasa interrumpe la transcripción de la secuencia de ADN a ARN. Con la transcripción bloqueada, la traducción del ARN en proteínas es imposible. Por lo tanto, los silenciadores previenen la expresión de genes a proteínas.
–Promotor: Los promotores contienen secuencias específicas de ADN, como los elementos de respuesta que son secuencias cortas de ADN donde se une la ARN polimerasa y proteínas llamadas “factores transcripcionales” (Ft) que se encargan de reclutar a la ARN polimerasa y regular la expresión génica.
–Región 5′ UTR: Esta región es importante para la regulación e iniciación de la transcripción a veces se traducen a un producto de aminoácidos. Este producto puede regular la traducción del ARNm.
–Marco de lectura abierto (ORF): El marco de lectura abierto es la parte de un gen que contiene el potencial de ser traducido a una proteína. Un ORF es un tramo continuo de codones que contienen un codón de inicio y de paro (por lo general AUG para el inicio y UAA, UAG o UGA como codones de paro).
–Codón de inicio: Es el primer codón del ARN mensajero transcrito (ARNm) traducido por un ribosoma. El codón de inicio siempre codifica para metionina en eucariotas y un Met modificado (fMet) en bacterias.
–Codón de paro: Es el codón de paro se encuentra dentro de una secuencia del ORF el cual indica la terminación de la traducción de la proteína.
–Región 3’UTR: La región 3’UTR es la sección de un gen que sigue inmediatamente a la traducción codón de terminación. Las regiones reguladoras de la región 3 ‘no traducida pueden influir poliadenilación, la eficiencia de traducción, localización, y la estabilidad del ARNm.
–Secuencia Terminador: Una secuencia terminador es una sección del gen que marca el final de un gen u operón durante la transcripción. Esta secuencia regula el término de la transcripción, proporcionando señales en que liberan el ARNm del complejo transcripcional, liberando la ARN polimerasa y la maquinaria transcripcional para comenzar una transcripción de nuevos ARNm.
–Exón: Un exón es cualquier parte de un gen que codifica una parte del ARNm. El término exón se refiere tanto a la secuencia de ADN dentro de un gen como a la secuencia correspondiente en las transcripciones de ARN. Durante el splincing del ARN, los intrones son eliminados y los exones se unen entre sí generando un ARN mensajero maduro. Al igual que el conjunto completo de genes para una especie constituye el genoma, todo el conjunto de los exones constituye el exoma.
–Intrón: Un intrón es cualquier secuencia de nucleótidos dentro de un gen que se elimina durante el splicing del ARN como parte del proceso de maduración del ARN. Los intrones se encuentran en los genes de la mayoría de organismos y muchos virus, y pueden estar situados en una amplia gama de genes. Los intrones no codifican productos de proteínas, pero son esenciales para la regulación de la expresión génica. Algunos intrones codifican ARN funcionales a través de procesamientos adicionales después de splicing generando moléculas de ARNs no codificantes.
*Mutaciones dentro de un gen: Durante la replicación del ADN, la división celular o por la exposición de agentes químicos o físicos en el medio ambiente pueden generar mutaciones. Las mutaciones son errores que surgen con frecuencia y para contrarrestarlos existen mecanismos de reparación celular que evitan que sean tan frecuentes o persistentes, sin embargo, cuando estos mecanismos fallan y el error en la lectura de la secuencia nucleotídica persiste se le llama se mutación. Las mutaciones pueden resultar en inserciones, deleciones, translocaciones entre otras, y pueden estos cambios producir modificaciones en la traducción, y por lo tanto, en el fenotipo de un organismo. Las mutaciones juegan un papel muy importante en los procesos biológicos normales y anormales, incluyendo la evolución de un organismo y/o el desarrollo de enfermedades.
Ingeniería genética
Desde la década de 1970 se han desarrollado técnicas para manipular a los genes. Estas técnicas han sido tan importantes que han permitido el desarrollo de una nueva rama de la biología llamada ingeniería genética, la cual permite la manipulación de los genes. Su objetivo principal es modificar los genes en un laboratorio e introducirlo en células vivas de la misma especie u otra diferente, de manera que pasa a formar parte de su genoma y logra así adquirir una nueva capacidad fenotípica. Su utilidad es la elaboración de productos que generalmente son nuevas proteínas. Estas técnicas tienen tanta importancia en la vida diaria y un ejemplo de esto es la producción de insulina humana en células de bacterias.
La ingeniería genética tiene aplicaciones en campos muy diversos; desde la medicina hasta la creación productos industriales como la producción de biocombustibles. El progreso en estos ámbitos puede aportar resultados capaces de solucionar grandes problemas de importancia mundial. Pero a pesar de su utilidad, su capacidad de mitigar y solucionar grandes problemas la ingeniería genética tiene graves problemas éticos en debate. Hay opiniones muy diversas sobre dónde deben de situarse los límites de la manipulación del material genético, que es la base de todos los procesos vitales.
Uno de los mayores exponentes de la ingeniería genética es la producción de la insulina, una proteína que ayuda a regular los niveles de azúcar en nuestra sangre. Normalmente insulina se produce en el páncreas, pero en personas con diabetes tipo 1 hay un problema con la producción de insulina. Por lo tanto, las personas con diabetes tienen que inyectarse insulina para controlar sus niveles de azúcar en la sangre. La ingeniería genética se ha utilizado para producir un tipo de insulina, muy similar a la nuestra, a partir de levaduras y/o bacterias como la E. coli .
Proceso de la ingeniería genética para la producción de insulina
Para facilitar la comprensión de cómo se lleva a cabo este procedimiento se resumirá en 8 pasos:
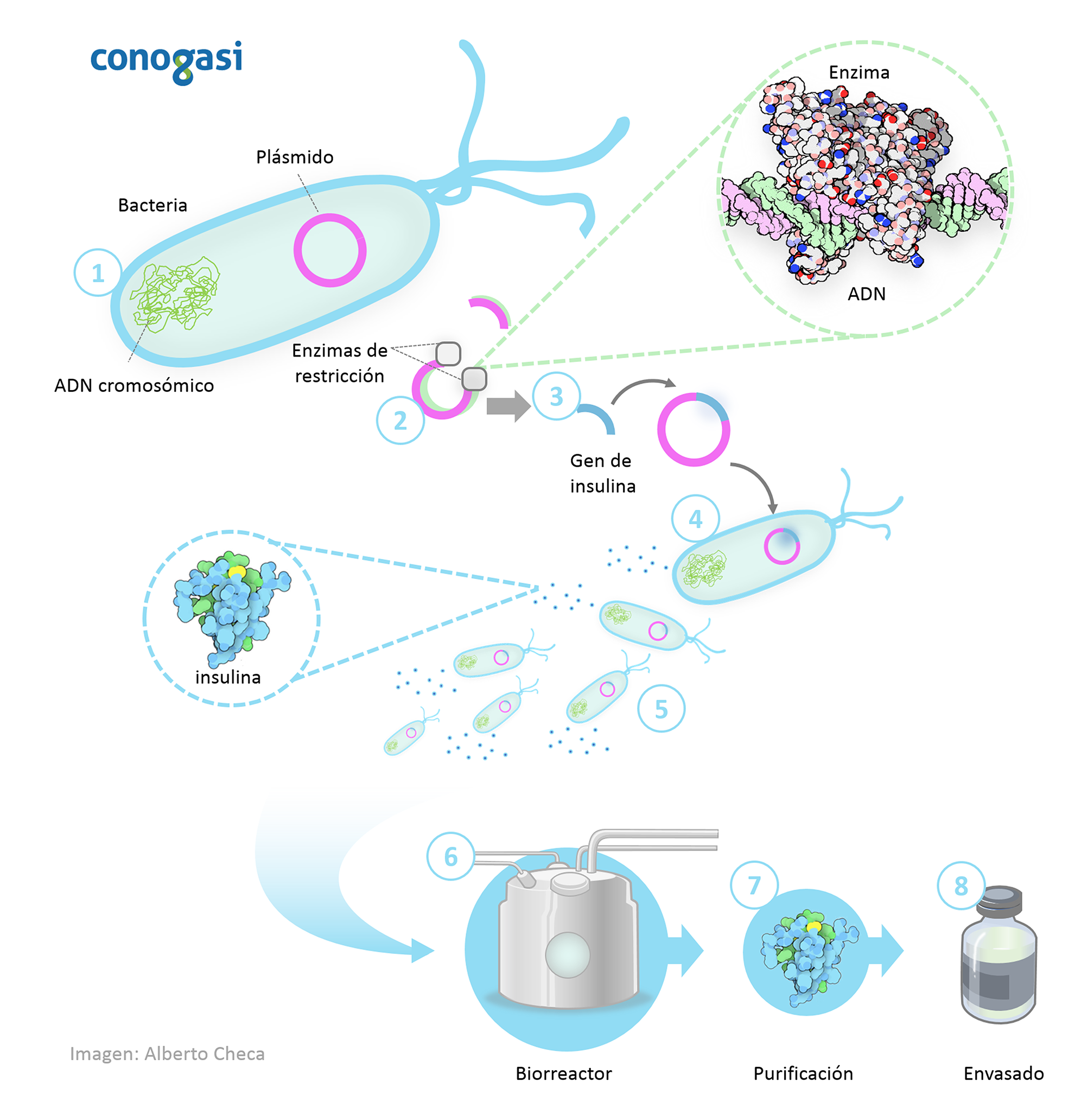
- Se toma ADN circular de origen microbiano llamado plásmido que puede extraerse de la célula de bacteria o levadura (generalmente bacteria).
- Luego se corta una pequeña sección del plásmido circular mediante enzimas de restricción, que son un conjunto de proteínas que pueden ser usadas como unas “tijeras moleculares”.
- Se toma el gen de la insulina humana diseñada con los sitios de corte de las enzimas para ser insertada en el espacio del plásmido. Cuando se ha colocado el gen de la insulina el plásmido ahora está genéticamente modificado.
- El plásmido genéticamente modificado se introduce a una nueva bacteria o célula de levadura.
- Esta célula se divide rápidamente y comienza a producir insulina.
- Para producir grandes cantidades de insulina, los microorganismos genéticamente modificados se cultivan en grandes recipientes de cultivo que contienen todos los nutrientes que necesitan, y cuanto más se dividen las células, más insulina se produce.
- Cuando se completa el crecimiento, la mezcla se purifica para liberar la insulina.
- La insulina se purifica y se envasa para su distribución a pacientes con diabetes.
Quieres saber más
Método: Extracción de ADN plasmídico (Lisis alcalina modificado)
Cómo citar: Checa Rojas, A. (2017, 09 de Mayo ) Gen: Desde el código genético hasta la ingeniería genética. Conogasi, Conocimiento para la vida. Fecha de consulta: Septiembre 18, 2025
Esta obra está disponible bajo una licencia de Creative Commons Reconocimiento-No Comercial Compartir Igual 4.0





Deja un comentario
Sé el primero en comentar!