Descripción
La PCR o reacción en cadena de la polimerasa es una técnica muy importante en el campo de la biología molecular y biotecnología para realizar clonación y manipulación de secuencias de ADN existentes. En este método se describe paso a paso cómo construir rápidamente un gen sintético (sin requerir un ADN molde) mediante una PCR de dos pasos (Dillon P.J. & Rosen C.A., 1990). Este método se basa en el uso de múltiples primers u oligonucleótidos solapantes para generar un gen o una fragmento de ADN sintético a través de varias rondas secuenciales de amplificación de PCR basada en Klenow. A diferencia de otros métodos como la clonación, este permite la generación de genes sintéticos en tan solo 1 día (Dillon P.J. & Rosen C.A., 1991).
Materiales
- Tubos para PCR
- Gradilla para tubos de PCR
- Micropipetas
- Guantes de nitrilo
- Puntas estériles y libres de nucleasas
- Termociclador
- Camara de electroforesis horizontal para ADN
- Fuente de poder
Reactivos
- Primers u oligonucleótidos que se sobrelapen (se requiere un diseño previo)
- Primers flanqueantes con sitios para las enzimas de restricción (opcional y se requiere diseño previo)
- Taq polimerasa
- 10X del Buffer de la PCR: 500 mM de KCl, 100 mM Tris-HCl, pH 8.0, 15 mM MgCl2.
- 10X dNTPs: 2 mM de cada dideoxinucleotido dATP, dCTP, dGTP y dTT.
- Agua ultra pura y libre de nucleasas
- Buffer TAE o TBE
- Agarosa
Procedimiento
- Antes de comenzar la PCR es necesario diseñar y pedir los primers (ver diseño de primers con SnapGene)
- Teniendo los oligonucleótidos listos, nos disponemos hacer la primera PCR
- Coloque en un tubo los siguientes reactivos:
Primera PCR.
| Buffer 10X para PCR | 10 μL |
| 10X dNTP | 10 μL |
| Oligonucleotidos | 0.5 μg |
| Taq polimerasa de ADN | 2.5 U |
| Agua ultra pura estéril y libre de nucleasas | llevar a 100 μL |
NOTA: Los cebadores flanqueantes no deberían incluirse en la primera PCR ya que dan como resultado la generación de muchos productos de diferentes tamaños que no amplifican en la segunda reacción de PCR.
|
Desnaturalización inicial |
94°C, 5 min |
| 10 ciclos |
94°C, 1 min (desnaturalización) |
|
55-60 °C, 1 min (alineamiento) |
|
|
72°C, 1 min (extensión) |
|
|
Extensión final |
72°C, 5 min |
5. Segunda PCR
| Buffer 10X para PCR | 10 μL |
| 10X dNTP | 10 μL |
| Primers flanqueantes | 1 μg |
| Taq polimerasa de ADN | 2.5 U |
| Templado de la primera reacción de PCR | 1 μL |
| Agua ultra pura estéril y libre de nucleasas | llevar a 100 μL |
|
Desnaturalización inicial |
94°C, 5 min |
| 25 ciclos |
94°C, 1 min (desnaturalización) |
|
55-60 °C, 1 min (alineamiento) |
|
|
72°C, 1 min (extensión) |
|
|
Extensión final |
72°C, 5 min |
7. Analice 10 μL de la 1° y 2° reacción de PCR mediante electroforesis en gel de agarosa. En la 1°reacción de PCR se observará un barrido y en la 2° reacción de PCR debe observarse una banda que corresponde al tamaño del producto deseado. NOTA: este protocolo es efectivo hasta secuencias sintéticas de 750 pb.
8. Esta banda corresponde a nuestro gen sintético y puede digerirse el producto de la 2° reacción de PCR para ser clonado en un vector adecuado.
NOTA: antes de clonar es aconsejable secuenciar el producto final para asegurar que la secuencia sea correcta. La tasa de error para este método debe aproximarse a la observada para otros protocolos de PCR que usan Taq polimerasa (ver hoja técnica de su fabricante).
Resultado
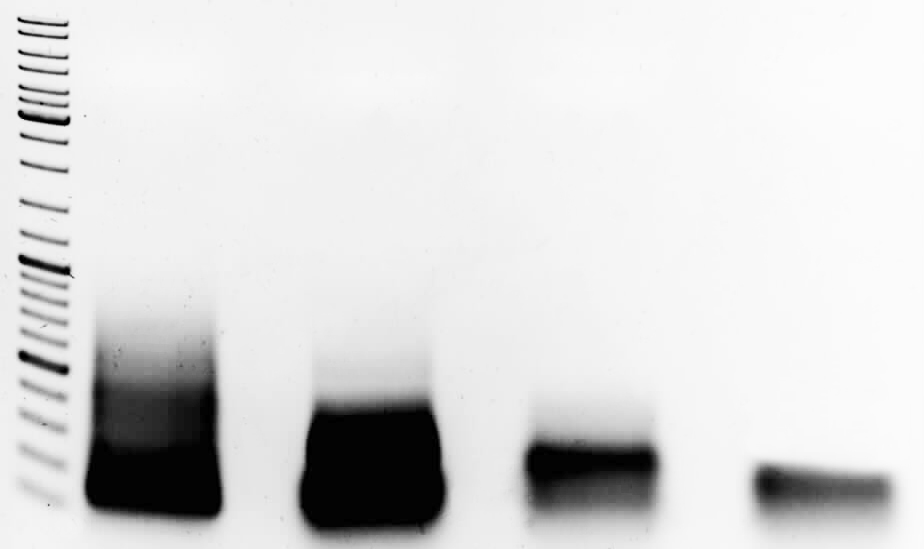
Cómo citar: Checa Rojas, A., Orlando Santillán Godínez (2018, 22 de Febrero ) Método: Rápida construcción de un gen sintético. Conogasi, Conocimiento para la vida. Fecha de consulta: Septiembre 4, 2025
Esta obra está disponible bajo una licencia de Creative Commons Reconocimiento-No Comercial Compartir Igual 4.0





Deja un comentario
Sé el primero en comentar!