Introducción
La reacción en cadena de la polimerasa (PCR) es una técnica sensible y muy útil para obtener y generar muchas copias de secuencias específicas de ADN de una muestra compleja de ADN con una diversidad y abundancia diferencial de secuencias; a este proceso se le conoce como: amplificación del ADN. Para llevar a cabo este proceso, se requiere de la utilización de una enzima llamada Taq Polimerasa que es ampliamente utilizada en estos métodos y sus variantes. Este método proporciona recomendaciones para garantizar una PCR exitosa.
Material
- Micropipetas
- Puntas para PCR
- Autoclave
- Tubos para PCR
- Gradilla para tubos de PCR
- Termociclador
Reactivos
- Buffer de Reacción Taq 10X
- dNTPs
- Primers
- ADN estándar
- Agua libre de Nucleasas
- Taq Polimerasa
Procedimiento
NOTA: Antes de comensar es recomendable preparar la reacción sobre hielo y precalentar el termociclador a 95 ° C. Todos los materiales deben estar esteriles (autoclave) y limpios.
- Prepare los tubos que habrá de utilizar para su PCR en una gradilla. NOTA: Preferentemente NO etiquete la tapa del tubo ya que se puede borrar, mejora etiquete en los costados
- Realice los cálculos de para preparar las reacciones necesarias
- Coloque en cada tubo los primers que ha de usar como el de la secuencia problema y control de carga ej.: B-actina, GAPDH, betaína, etc.
- Agregue cada componente de la tabla 1 en orden descendente en un tubo y marque el tubo como MasterMix. NOTA: coloque la polimerasa al final. Saque del congelador la enzima y regresela inmediatamente después.
- Coloque una cantidad proporcional en cada tubo y pongola en el termociclador (figura 1) ya programado con las condiciones de la PCR (ver tablas abajo).
- Al terminar su PCR analice su producto en un gel de agarosa.
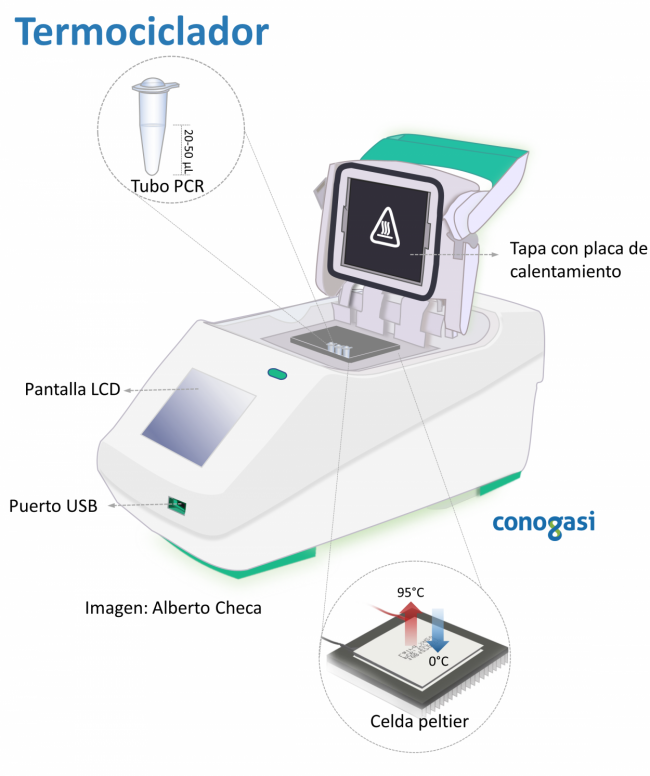
Tabla 1. Reactivos para la PCR
| Reactivos | 25 μL para 1rx | 50 μL para 1rx | Concentration Final |
| Buffer de Reacción Taq 10X | 2.5 μL | 5 μL | 1X |
| dNTPs 10 mM | 0.5 µL | 1 μL | 200 µM |
| Primer 3′ 10 µM | 0.5 µL | 1 μL | 0.2 µM (0.05–1 µM) |
| Primer 5′ 10 µM | 0.5 µL | 1 μL | 0.2 µM (0.05–1 µM) |
| ADN molde | variable (entre 2-10 µL) | variable | <1,000 ng |
| Agua libre de Nucleasas | lleve a 25 µL | lleve a 50 µL | |
| Taq Polimerasa | 0.125 µL | 0.25 µL | dependiendo del proveedor |
NOTA: La amplificación de ADN molde estructura secundaria alta, alto contenido de GC, bajas concentraciones o con más de 5 kb puede requerir una mayor optimización.
Condiciones del Termociclador para una PCR convencional
| Pasos |
Temperatura |
Tiempo |
| Desnaturalización | 95°C | 1 minuto |
| 25-30 Ciclos | 95°C 45-68°C 68-72°C |
15-30 segundos 15-60 segundos 1 minute/Kb |
| Extensión Final | 68-72°C | 5 minutos |
| Mantener a | 4-10°C | ∞ |
PCR de dos pasos: 2-step PCR
Cuando los primers tienen una temperatura de alineamiento superior a 65°C se debe realizar una PCR de dos pasos.
Condiciones de la PCR para un protocolo de PCR de 2 pasos:
| Paso | Temperatura |
Tiempo |
| Desnaturalización Inicial | 95°C | 30 segundos |
| 25-30 Ciclos | 95°C 65-68°C |
15-30 segundos 1 minutos/Kb |
| Extensión Final | 65-68°C | 5 minutos |
| Temperatura Final Mantener a | 4-10°C | ∞ |
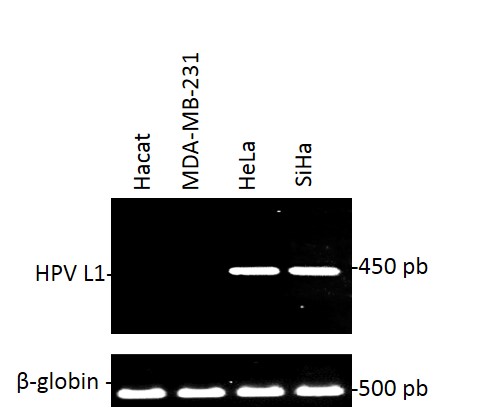
Resultado
Cómo citar: Checa Rojas, A. (2017, 14 de Noviembre ) Método: PCR. Conogasi, Conocimiento para la vida. Fecha de consulta: Octubre 19, 2025
Esta obra está disponible bajo una licencia de Creative Commons Reconocimiento-No Comercial Compartir Igual 4.0





Deja un comentario
4 Comentarios en "Método: PCR"
¿Necesitan sacar la sangre para la prueba de pcr?
Se puede sacar de cualquier muestra biológica que tenga ácidos nucleicos es decir: órganos, tejidos y células. Pero también se puede obtener de muestras que se sepa que existe ADN o ARN.
Se pueden utilizar los mismos primers para PCr tiempo real y Pcr punto final o tienen que ser especificos para cada prueba