Resumen
La edición genómica son técnicas que usan herramientas de biología molecular para realizar cambios en el ADN del genoma de una célula o un organismo como: agregar, eliminar, modificar o alterar nucleótidos o secuencias. Enzimas de diseño cortan el ADN en una secuencia específica, y cuando la célula lo repara, se realiza un cambio o se edita la secuencia cortada. La edición del genoma no es una técnica o un concepto nuevo se ha utilizado desde hace años para modificar con precisión y eficacia el ADN haciendo cortes en secuencias de ADN específicas usando nucleasas acopladas a dedos de zinc o efectores de tipo activador de la transcripción. Recientemente se ha desarrollado una técnica llamada CRISPR Cas9, ésta técnica se ha convertido en una revolución por sus futuras implicaciones en la microbiología, pero, se considera aún más revolucionario debido a que es mucho más fácil en su procedimiento, y por su mayor grado de precisión que sus antecesores. CRISPR Cas podría llevarnos a un cambio de paradigma desde la como manipulamos un genoma hasta como la forma en que concebimos a la vida misma. Esta técnica nos abre las puertas para hacer investigación en el campo de genómica humana que permita revolucionar su aplicación en la terapia génica. El potencial terapéutico se está aprovechando para tratar trastornos monogénicos como la distrofia muscular y la hemofilia, y se llega a considerar como una mina de oro farmacéutica valuada en mil millones de dólares; esto se evidencia por el capital que se invierte para expandir sus alcances médicos; ya que, el que domine esta técnica en células dominará la industria farmacéutica.
Introducción
Edición genómica
La edición del genoma es la inserción, eliminación o el reemplazo de secuencias especificas de ADN dentro de un genoma de un organismo o una célula. Generalmente está técnia se realiza en un laboratorio usando enzimas de diseño llamadas nucleasas. La edición genómica no es un concepto nuevo y CRISPR Cas9 (el más famoso hoy en día) no fue el primer método de edición de genes. Antes de CRISPR Cas9, se descubrieron otros métodos de edición de genes, como los dedos de zinc (ZFNs), las nucleasas efectoras de la transcripción de tipo activador (TALENs), Virus adeno-asociado recombinante (rAAV, por sus siglas en ingles) y transposones; estos dos últimos no será abordados en este árticulo (Joung & Sander, 2013; Miller et al., 2007; Urnov, Rebar, Holmes, Zhang, & Gregory, 2010; Wood et al., 2011).
ZFNs
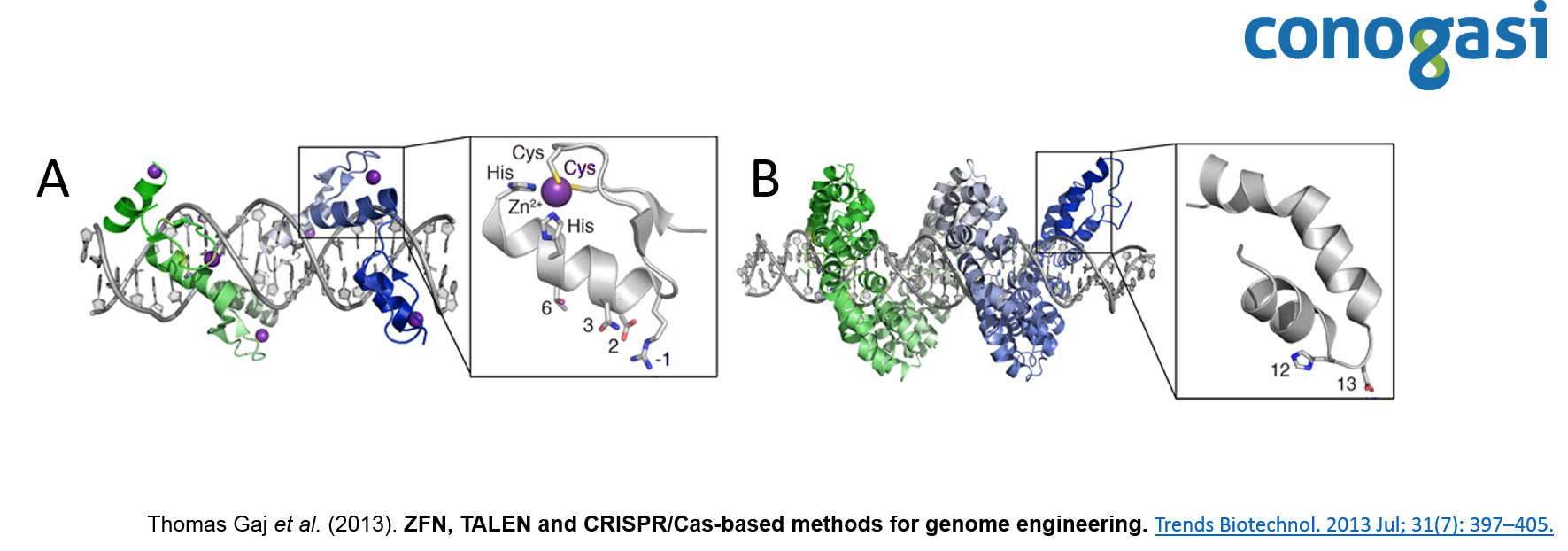
Las nucleasas con dedo de zinc (ZFNs) son enzimas de restricción sintéticas generadas con un dominio de unión al ADN mediante dominio de dedo de zinc, que reconoce un sitio de corte específico de ADN. El dominio de dedo de zinc Cys 2 -His 2 se encuentra entre los tipos más comunes de motivos de unión de ADN encontrados en organismos eucariotas y representa el segundo dominio de proteína con mayor frecuencia en el genoma humano. Un dedo de zinc individual consta de aproximadamente 30 aminoácidos en una configuración conservada de hojas beta-beta/alfa hélices (ββα) (figura 1). Generalmente, varios aminoácidos de la α-hélice tienen contacto con tres pares de bases (bps) del ADN, con diferentes niveles de selectividad (Thomas Gaj et al. 2013). Los dominios de dedos de zinc se diseñan para cortar secuencias específicas de ADN y se aprovecha la maquinaria endógena de reparación del ADN, para modificar con precisión secuencias dentro de los genomas de los organismos superiores. Debido a su funcionalidad se convirtió en una técnica atractiva para el diseño de blancos personalizados de ADN.
TALEN
Por otra parte, las proteínas efectoras de tipo activador de la transcripción (TALE) están compuestas por un dominio central responsable de la unión al ADN, una señal de localización nuclear y un dominio que activa la transcripción del gen blanco (Schornack, Meyer, Römer, Jordan, & Lahaye, 2006). El dominio de unión al ADN consiste en monómeros, cada uno de ellos se une a un nucleótido en la secuencia de nucleótidos diana. Los monómeros son repeticiones en tándem de 34 residuos de aminoácidos, dos de los cuales se localizan en las posiciones 12 y 13 y son altamente variables (figura 1), y son ellos los responsables del reconocimiento de un nucleótido específico (Lamb, Mercer, & Barbas, 2013). Después de descifrar la secuencia de ADN de las proteínas TALE, atrajo la atención de muchos investigadores debido a su simplicidad (un monómero – un nucleótido), y se publicaron los primeros estudios sobre la construcción de nucleasas quimérica TALEN (Christian et al., 2010). Esto dio como resultado la generación de numerosas construcciones genéticas que expresan las nucleasas quiméricas artificiales con el dominio de unión al ADN y el dominio de la endonucleasa FokI. Lo que permitió este sistema, combinando con monómeros del dominio de unión al ADN, construir nucleasas artificiales, cuyo objetivo puede ser cualquier secuencia de nucleótidos. Después de descifrar la secuencia de ADN de las proteínas TALE, trajo la atención de muchos investigadores debido a su simplicidad (un monómero – un nucleótido), y se lanzaron los primeros estudios sobre la construcción de nucleasas quimérica TALEN (Christian et al., 2010). Esto dio como resultado la generación de construcciones genéticas que expresan las nucleasas quiméricas artificiales con el dominio de unión al ADN y el dominio de la endonucleasa FokI. Este sistema permite, combinando monómeros del dominio de unión al ADN con diferentes RVD, construir nucleasas artificiales, cuyo objetivo puede ser cualquier secuencia de nucleótidos (Nemudryi, Valetdinova, Medvedev, & Zakian, 2014).
CRISPR-Cas 9
Descubrimiento de CRISPR
La edición de genes, basada en una tecnología conocida como CRISPR-Cas9 fue descubierta por Francisco Mojica en 1993, junto con su asesor. Estos investigadores se encontraban estudiando la arquea Haloferax mediterranei, la cual tiene la característica de tolerar la salinidad y afectar a las enzimas de restricción. Mojica encontró múltiples copias de una secuencia palindrómica de 30 bases, separadas por espaciadores de aproximadamente 36 bases que no se parecían a ninguna familia de repeticiones conocidos en microorganismos (Lander, 2016; F. J. Mojica, Juez, & Rodríguez-Valera, 1993). Pronto descubrió estructuras similares en microorganismos distantes que indicaban una función en procariotas, a esta enzima la nombro SRSR’s (short regularly spaced repeats) y fue renombrada después como CRISPR (clustered regularly interspaced short palindromic repeats) (Jansen, Van Embden, Gaastra, & Schouls, 2002; F. J. M. Mojica, Ferrer, Juez, & Rodríguez‐Valera, 1995).
En busca de la función de CRISPR
Mojica propuso una serie de hipótesis para la función de CRISPR como la regulación génica, la replicación y en la reparación del ADN; pero sin evidencias experimentales (Mojica y Garrett, 2012). En un intento de demostrar su función, encontró una similitud con una secuencia de un fago, llamado P1. Este fago infecta a muchas cepas de E. coli, y esta secuencia se encontraba en un locus de una cepa de E. coli que recientemente secuenció y era resistente a la infección de P1. Basándose en esos resultados, analizó más espaciadores y la mayoría estaban relacionados con secuencias virales o con plásmidos de conjugación. Mojica se dio cuenta de CRISPR debían codificar las instrucciones para un sistema inmune adaptativo que protegía a las bacterias contra ADN ajeno. Mojica publicó estos resultados en el Journal of Molecular Evolution (F. J. M. Mojica, Díez-Villaseñor, García-Martínez, & Soria, 2005), donde se informó la posible función de CRISPR, pero antes de publicar estos resultados fue rechazado de las revistas Nature, PNAS y Molecular Microbiology y Nucleic Acid Research argumentando que no contaba con la suficiente relevancia o incluso que esto ya se conocía (Lander, 2016). Por otra parte, Christine Pourcel y sus colaboradores publicaron un mes después el posible papel del locus CRISPR en Y. pestis donde proponen que estos repetidos podrían servir como una defensa ante posibles ataques contra bacteriófagos y esta secuencias podrían servir además, como una posible herramienta de identificación (Pourcel, Salvignol, & Vergnaud, 2005).
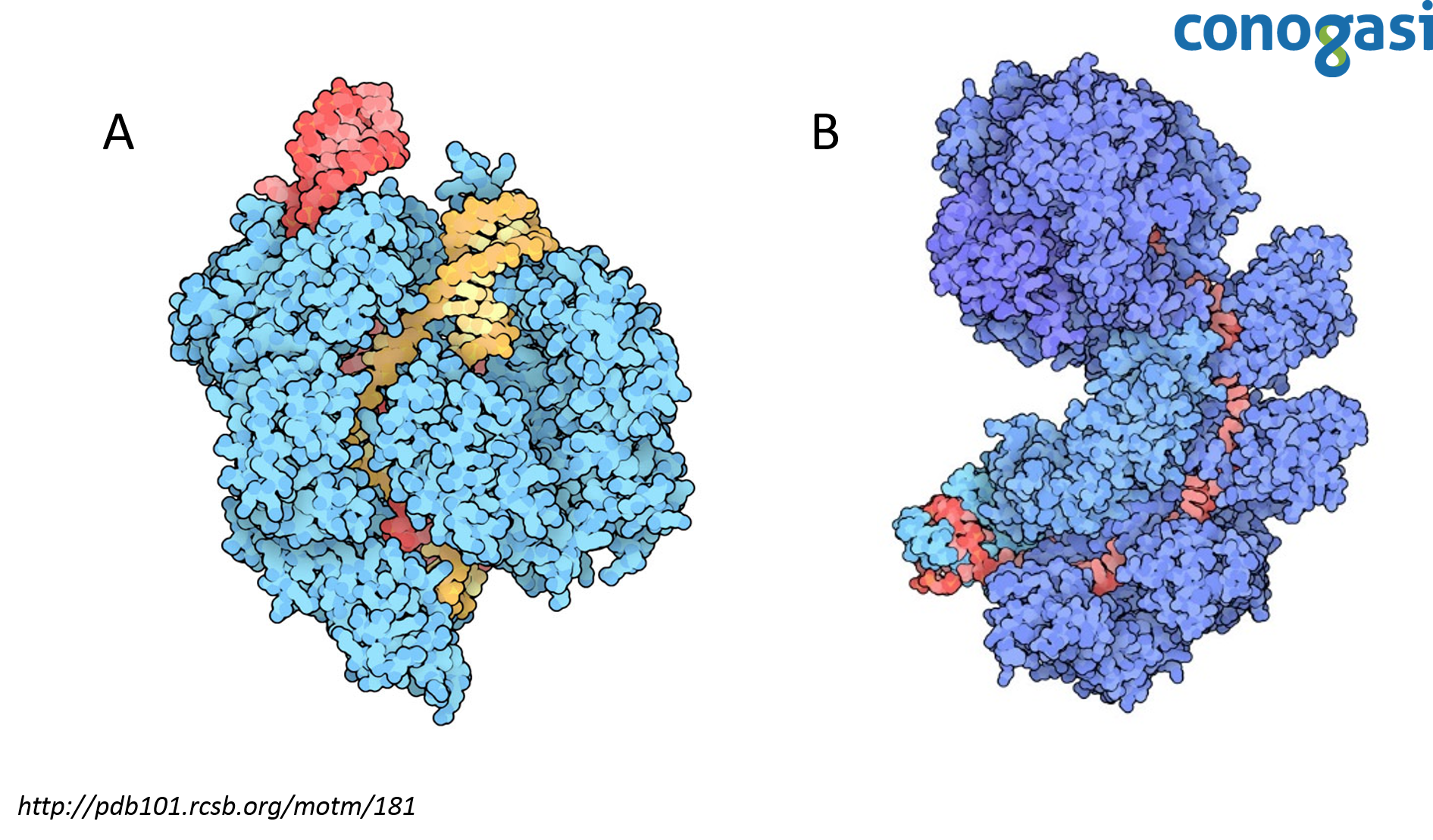
En estudios con cepas resistentes a la infección contra fagos en S. thermophilus, se encontró que en lugar de albergar mutaciones clásicas en los receptores de superficie celular que son necesarios para la entrada del fago, las bacterias habían adquirido secuencias derivadas de los fagos en los loci de CRISPR. Por otra parte, se estudió el papel de las proteínas relacionadas a CRISPR, las proteínas Cas7 y 9 (figura 2). Las bacterias usan Cas7 para obtener la resistencia y por otro lado, la proteína Cas9 contiene dos motivos de nucleasas necesarias para la resistencia contra los fagos y es el componente activo en el sistema inmune bacteriano. Los fagos que superan la defensa de CRISPR realizan cambios en sus genomas alterando la secuencia correspondiente al espaciador ya que no se lleva a cabo el reconocimiento de CRISPR porque depende de una secuencia precisa de ADN (Bolotin, et al., 2005; Makarova, et al. 2006).
Aplicación de CRISPR
En el 2008 el científico van der Oost y sus colaboradores introdujeron un sistema CRISPR de E. coli en otra cepa de E. coli carente del sistema endógeno, esto les permitió caracterizar un complejo de cinco proteínas Cas, llamada complejo Cascada (Brouns et al., 1993). El complejo cascada requiere de las cinco proteínas Cas unidas a un complejo de ARN/CRISPR (crRNA). Van der Oost y su equipo realizó una inactivación (knockout) de cada componente y observaron que el complejo cascada es necesario para cortar un precursor largo de ARN que es transcrito desde el locus de CRISPR de 61 nucleótidos de longitud (CRISPR ARN crRNAs) (figura 2) (Sorek et al., 2008). Para demostrar que las secuencias de crRNA son responsables de la resistencia basada en CRISPR, se programó a CRISPR para dirigirse a cuatro genes esenciales en el fago lambda (λ). Las cepas mostraron resistencia al fago λ y este fue el primer caso de programación directa de la inmunidad basada en CRISPR. Los resultados sugirieron también que el objetivo de CRISPR no es sobre el ARN sino sobre el ADN. Aunque los resultados iban dirigidos a ambos, estos variaron en su eficacia, pero los resultados sugerían que el objetivo no era mRNA. Sin embargo, esta evidencia fue indirecta, quedando solo como una hipótesis de que CRISPR se dirigía al ADN (Lander, 2016).
Tras confirmar que CRISPR es un mecanismo del sistema inmune adaptativo en bacterias, era necesario entender el mecanismo de corte que CRISPR hacía en el ADN. Debido a la gran eficiencia de CRISPR para eliminar el ADN de un invasor no era fácil observar este proceso. Para resolver este problema encontraron un conjunto de cepas de S. thermophilus en las que CRISPR confería una protección parcial y el proceso se había ralentizado lo suficiente para observar los productos directos de la acción de CRISPR. Con estas cepas, pudo observar que CRISPR dependía de la nucleasa Cas9 (figura 2). Estos resultados demostraron la actividad de nucleasa de Cas9 actuando sobre el ADN en posiciones precisas codificadas por los crRNAs (Barrangou et al., 2007; Garneau et al., 2010).
Mecanismos de CRISPR
Tratando de identificar moléculas de ARN de microorganismos, Emmanuelle Charpentier y Jörg Vogel, descubrieron un ARN pequeño llamado ARN trans-activador (tracrRNA). Analizaron mediante herramientas de bioinformática regiones intergénicas para hallar estructuras secundarias que pudieran codificar para ARNs no codificantes y encontraron regiones candidatas las cuales incluía una región cercana al locus de CRISPR. Utilizando secuenciación masiva, observaron que la tercera clase de transcrito más abundante (después del ARNr y el ARNt) fue un ARN pequeño que se transcribe a partir de una secuencia adyacente a CRISPR. Este ARN pequeño complementaba casi a la perfección con los repetidos de CRISPR. Diferentes experimentos demostraron que el tracrRNA era esencial para el procesamiento del crRNAs y, por tanto, para la función de CRISPR (Deltcheva et al., 2011). Estudios posteriores revelaron que el tracrRNA tiene otro papel fundamental, y que, no sólo participa en el procesamiento del crRNA, sino que es esencial para el complejo de nucleasa Cas9 al cortar el ADN (Jinek et al., 2012, Siksnys et al., 2012).
Posteriormente, Rimantas Sapranauskas y sus colaboradores demostrando que Cas9 es la única proteína necesaria para la interferencia y que sus dominios RuvC- y HNH-nucleasa son esenciales para la interferencia. Adema demostraron que el sistema CRISPR Cas puede ser clonado de una bacteria a otra proponiendolo ya como un sistema para reforzar la inmunidad en bacterias (Sapranauskas et al., 2011). Por otra parte, y de manera independiente la Dra. E. Charpentier y la Dra. Jeniffer Dounda utilizaron un sistema recombinante de S. pyogenes Cas9, además de un crRNA y tracrRNA que se expresaba en E. coli y observaron que el ADN podía cortarse in vitro y podía editarse mediante un diseño personalizado (Jinek et al., 2012).
CRISPR en humanos
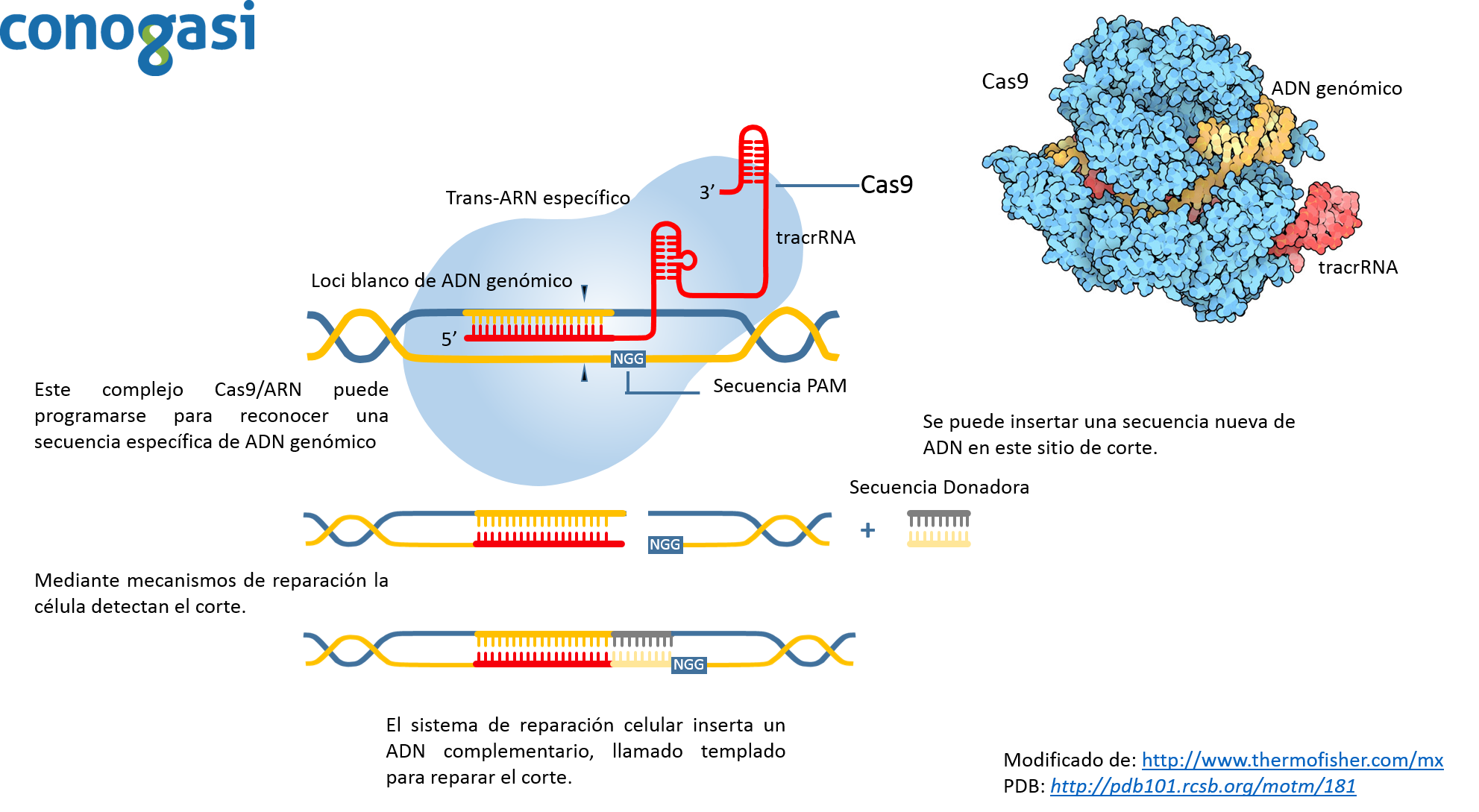
El método de CRISPR es revolucionario por sus implicaciones en la microbiología, pero se considera aún más revolucionario debido a que es mucho más fácil su procedimiento y por su mayor grado de precisión que sus antecesores. Debido a esto, el investigador Zhang y sus colaboradores (con experiencia en los antecesores de CRISPR en células de mamíferos) decidieron aplicar esta técnica y aplicarla en células de mamífero (Lander, 2016; Zhang et al., 2011). Probaron diferentes enzimas de Cas9 y observaron que sus eficiencias eran diferentes, encontrando que la enzima Cas9 de S. pyrogenes como la más eficiente. Además, de probar diferentes isoformas de tracrRNA para identificar la más estable en células humanas. En el 2012 lograron bajo un sistema de tres componentes (CRISPR, Cas9 de S. pyrogenes o S. thermophilus y un tracrRNA) orientar la edición de genes en genomas de humano y de ratón con una alta eficiencia mediante la recombinación no homologa (Lander, 2016).Aunque la tecnología de edición basado en la endonucleasa CRISPR Cas9 está permitiendo una amplia gama de aplicaciones, como la modificación de genes de diferentes organismos como: plantas, ganado y organismos modelo de laboratorio (roedores, pez zebra, etc.) (Hsu, Lander, & Zhang, 2014). Cas9 puede ser guiado a lugares específicos dentro de genomas mediante un ARN guía (figura 3). Usando este sistema, las secuencias de ADN dentro del genoma endógeno y sus salidas funcionales están ahora editados o modulados en prácticamente cualquier organismo de elección. Y ya que el procedimiento de edición genética es simple y escalable, ha permitido a los investigadores dilucidar la organización funcional del genoma en el nivel del sistema y establecer vínculos causales entre las variaciones genéticas y los fenotipos biológicos. La corrección de los errores genéticos asociados con enfermedades sugiere que la edición tiene aplicaciones potenciales en la terapia génica para los seres humanos y la industria.
Los primeros experimentos en humanos
En 2015 se realizaron los primeros experimentos en células embrionarias humanas tratando de modificar el gen responsable de la β-talasemia, un trastorno sanguíneo potencialmente mortal. Los investigadores reportan en sus resultados que existen serios obstáculos para utilizar el método en aplicaciones médicas. Ya que, solo 4 de los 54 embriones que usaron se logró la edición del gen, pero estos eran mosaicos, esto quiere decir que, no todas sus células fueron eficientemente editadas; además, de que se provocaron mutaciones en genes relacionados. Los resultados son tan poco alentadores que, se requieren de modificaciones y más estudios para mejorar aún esta tecnología (Liang et al., 2015). Por otro lado, el comité internacional convocado por la Academia Nacional de Ciencias de Estados Unidos (NAS por sus siglas en ingles), han aprobado el estudio en embriones humanos bajo ciertas circunstancias, y después de analizar rigurosamente los riesgos/beneficios, han limitado el uso solo a parejas donde la edición sea estrictamente necesaria para concebir un hijo biológicamente sano. Según el investigador Erick Lander debido a las condiciones éticas y legales impuestas, esto es muy alentador para un conjunto muy particular de investigadores, pero desalentador para otros, debido a las limitaciones impuestas esto solo permite que un grupo muy reducido de investigadores puedan realizar este tipo de investigaciones. Aunque el debate aún continúa, existen opiniones con tendencias marcadas hacia los intereses económicos (Kaiser, 2017). Y esto es obvio, ya que, el que domine la edición genética sobre las células humanas especialmente sobre la línea germinal, adquirirá el control sobre el mercado farmacéutico mundial.
Por otro lado, otro aspecto decepcionante de esta gran revolución médico-científica es la intervención de las “Big-Pharma”, ya que, si bien están haciendo grandes aportaciones financieras en pro del avance científico y tecnológico de la edición genómica, tienen un objetivo muy claro que es recuperar su inversión a como dé lugar en el menor tiempo posible. Un problema es que las nuevas terapias que utilizan la edición genómica (ya sea, TALEN, ZFN o CRISPR), están enfocadas a enfermedades monogénicas o donde la incidencia de la enfermedad no es muy alta y, por lo tanto, el número de pacientes que usen estos tratamientos es muy reducido. Por lo que, algunos analistas de estas big-pharmas están calculando que estos tratamientos puedan tener costos entre $500,000 hasta el millón de dólares. Recientemente, se ha dado los primeros ejemplos de los altos costos que podrán alcanzar estas terapias, como la terapia Glybera que está dirigida para una enfermedad ocular mediada por el gen RPE65 (aprobada en China y Europa) con un costo estimado de $500,000 USD por ojo. Otro tratamiento, llamado Kymriah recientemente aprobado por la FDA (más información vea: Farmacogenómica: hacia una revolución…) y el primero en su tipo, está dirigido a pacientes con leucemia y tiene un costo de $475,000 dólares por paciente (E. Mullin, 2017, Checa Rojas, 2017). Si bien, las agencias reguladoras están haciendo un esfuerzo para actualizar su regulación sobre la aplicación de estos tratamientos, aún están muy lejos de regular los altos costos de la aplicación de estas terapias (Juliet Preston, 2017).
Conclusión
El progreso en el estudio de la edición genómica ha hecho evidente que será capaz de aportar y solucionar grandes problemas mundiales como salud, alimentos, procesos industriales, entre otros. Pero a pesar de su utilidad y su capacidad para poder mitigar o resolver estos problemas, existen actualmente argumentos éticos y legales que están en debate. Existen opiniones muy diversas sobre dónde han de situarse los límites de la manipulación del material genético, que es la base de todos los procesos vitales. Ya que, el que controle la manipulación genómica, podrá definir el destino de la vida humana y, por lo tanto, dominar la industria farmacéutica mundial.
Cómo citar: Checa Rojas, A. (2017, 19 de Octubre ) CRISPR-Cas: la nueva edición genómica y la búsqueda del dominio farmacéutico. Conogasi, Conocimiento para la vida. Fecha de consulta: Octubre 2, 2025
Esta obra está disponible bajo una licencia de Creative Commons Reconocimiento-No Comercial Compartir Igual 4.0





Deja un comentario
3 Comentarios en "CRISPR-Cas: la nueva edición genómica y la búsqueda del dominio farmacéutico"
Se ve que le echaron ganas a hacer este artículo. Sin embargo, no hay una sola frase, al inicio, que explique a quien no sabe, esto de qué se trata, porque no se explica en palabras diáfanas qué quiere decir "editar el genoma" o el ADN. Y dos, está a mi gusto muy largo, lo que habrá que baje la frecuencia de que lo lean todo. Recomiendo hacer al menos dos artículos de este,, otro, con el mismo título y agregando "Resumen de.." que sea la mitad o incluso menos largo que este. Saludos
Sería padre que no faltara un solo acento. Dices: " Estudios con cepas resistentes a la infección contra fagos en S. thermophilus, se encontró que…" la sintaxis está mal. Se corrige diciendo: "En estudios con cepas resistentes a la infección contra fagos en S. thermophilus, se encontró que…"
Creo que el artículo no menciona nada de la segunda parte de su título: …y la búsqueda del dominio farmacéutico?? Se ve que el que escribe el texto tiene muchas ideas en su cabeza, pero no se da cuenta que tal vez no las ha escrito. Suele pasar