Conocimientos previos
- ADN
- Extracción del ADN
- Síntesis de ADNc
Descripción
La PCR o la reacción en cadena de la polimerasa (por sus siglas en inglés) es un método por el cual se obtiene gran número de copias de un fragmento de ADN a partir de una muestra llamada ADN molde. Este método se basa en la función que tienen unas proteínas llamadas polimerasas, que sirven para duplicar el ADN durante la división celular. En general la PCR emplea ciclos de temperaturas altas (95°C) para separar las cadenas de ADN y posteriormente se disminuye la temperatura para duplicar nuevas cadenas de ADN. La PCR es un método ampliamente usado en las áreas biológicas, porque en la investigación es necesario utilizar un gran número de copias de ADN para su estudio y análisis. En estudios biomédicos se usa para el diagnóstico de enfermedades.
Materiales
Notas: Todos los materiales que se usan para la PCR deberán estar químicamente limpios y estériles, con el fin de evitar la degradación del ADN. Las copias de ADN tendrán que ser observadas mediante un gel de electroforesis.
- ADN molde: secuencias de ADN, provenientes del material biológico de nuestro interés y que se desea generar muchas copias
- Iniciadores o “primers“: son dos secuencias cortas de nucleótidos que son complementarias al fragmento del ADN molde. Están constituidas generalmente entre 20-40 nucleótidos en promedio y con un contenido de GC de 40-60%. El diseño o el análisis de estos puede realizarse en programas computacionales como SnapGene. La concentración final de cada primer pude ser de 0.05–1 μM.
- dNTPs: son nucleótidos que sirven para generar las nuevas cadenas de ADN. La concentración de los dNTPs es de 200 μM de cada deoxynucleótido.
- Polimerasa: es un tipo de enzima que duplica el ADN molde
- Iones: son necesarios para la función de la polimerasa, se usa generalmente iones de magnesio y están en forma de sal de cloruro de magnesio MgCl2 .La concentración final de Mg++ debe estar entre 1.5-2.0 mM y es óptima para la mayoría de los productos de PCR. Sin embargo, la concentración de Mg puede optimizarse incrementando de 0.5 o 1.0 mM. La amplificación de secuencias ricas en GC, puede mejorarse con betaína, DMSO o formamida.
- Termociclador: aparato que automatiza la PCR, eleva y baja la temperatura en tiempos cortos para poder llevar a cabo la reacción mediante una celda peltier (Figura 1).
- Ciclos de PCR: generalmente son de 25 a 35 ciclos, suficientes para obtener un producto de PCR (multiples copias de una secuencia en específico). Cuando se tiene un blanco con un bajo número de copias se requeriran arriba de 45 ciclos.
- Desnaturalización: inicialmente la PCR comienza con un ciclo de 2 a 4 minutos a 95°C para romper completamente la estructura secundaria del ADN molde. ¨Para PCR provenientes de secuencias ya amplificadas, la desnaturalización inicial se recomienda solo con 30 segundos a 95°C. Para la PCR de colonia una desnaturalización inicial de 5 minutos es recomendada.
- Alineamiento: durante este paso la temperatura disminuye y está basada en la temperatura de fusión de los primers (Tm) y puede estar entre 45 a 68°C. Las temperaturas de Tm se pueden optimizarse haciendo una PCR con gradiente de temperatura en 45°C e ir subiendo gradualmente cada 5 °C por debajo de la Tm calculada. Esta temperatura tiene una duración entre 15 a 60 segundos. Existen varios programas para calcular una temperatura de alineamiento o Tm apropiada como: SanpGene, Thermo, Biophp, etc.
Cuando los primers se alinean a temperaturas por arriba de 65°C, es recomendable realizar una PCR de 2-pasos (ver: Método de PCR). - Extensión: la temperatura de extensión recomendada es de 68-72°C. Los tiempos de extensión generalmente son de 1 minuto por kilo base (Kb). Se recomienda una extensión final de 5 minutos a 68°C. Si la Tm de los primers esta a 5°C cercano a la temperatura de extensión (68-72°C), considere ejecutar un protocolo de PCR de dos pasos. NOTA: La tempera de la Tm no debe exceder la temperatura de extensión.
- Ciclos de PCR: generalmente son de 25 a 35 ciclos, suficientes para obtener un producto de PCR (multiples copias de una secuencia en específico). Cuando se tiene un blanco con un bajo número de copias se requeriran arriba de 45 ciclos.
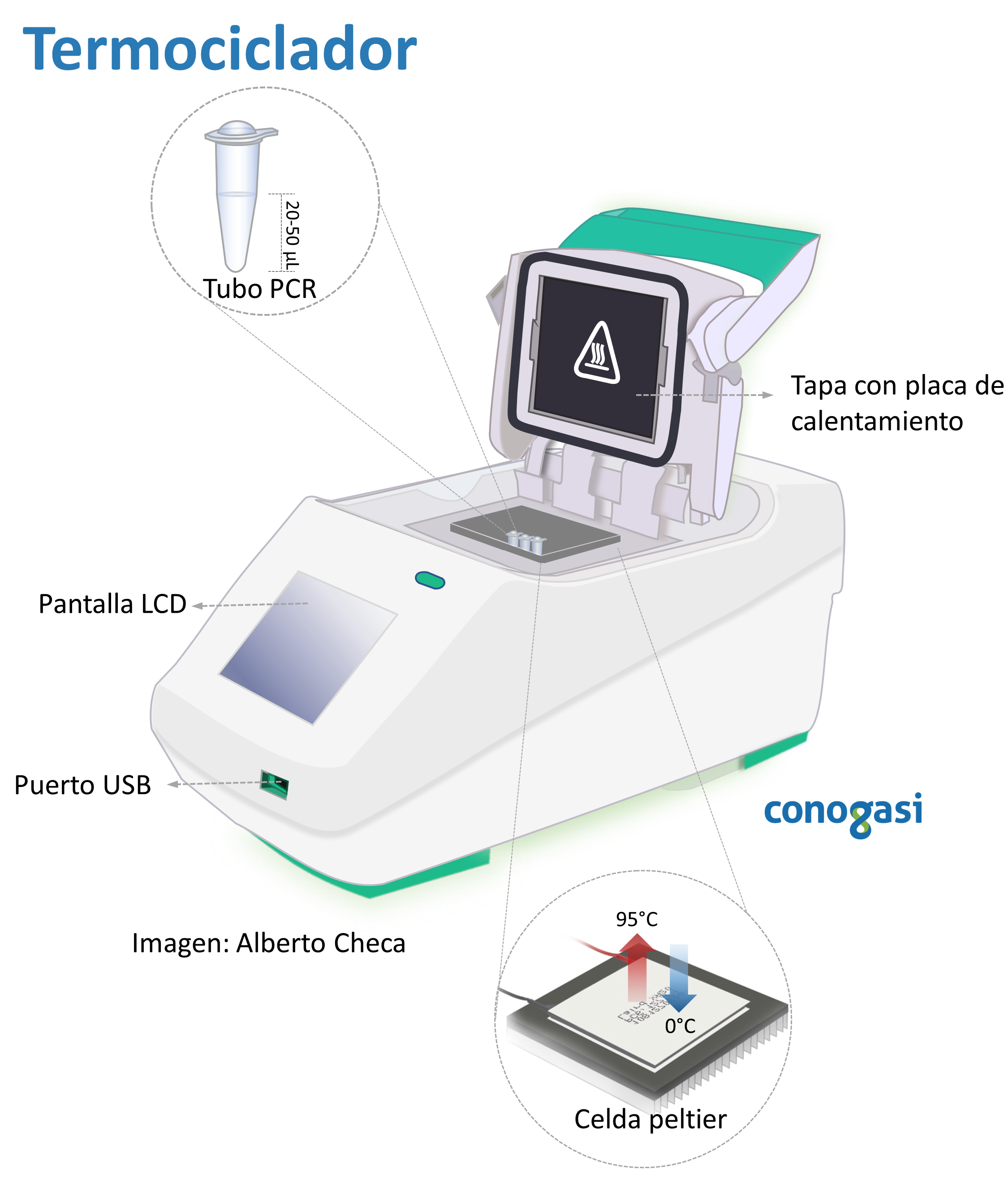
Requisitos previos
- Extracción de ADN
- Extracción de ARN
- Aislamiento de ARNm y síntesis de ADNc.
Procedimiento
- Mezcla maestra: es la mezcla de todos los componentes para la PCR colocados en un recipiente (ADN molde, iniciadores, dNTPs, iones y polimerasa) y se coloca en el termociclador
- Desnaturalización del ADN molde: se separa el ADN molde en dos cadenas de ADN, al calentar el recipiente de la mezcla maestra a 95°C por 5 minutos aproximadamente
- Alineación: se baja la temperatura a un rango de 55-65°C para que se unan los iniciadores al ADN molde y comience la duplicación del ADN
- Extensión: la polimerasa termina de duplicar el ADN molde formando nuevas cadenas de ADN a 72°C
- Ciclo de PCR: terminando la extensión se repite todo el proceso (30-35 veces)
- Cuantificación: proceso por el cual se mide la concentración del ADN de la PCR
Resultado
Múltiples copias de ADN provenientes de un fragmento ADN deseado. ¿Quieres saber cómo hacerlo?: Método: PCR
Fuentes de error más frecuentes
- Mala purificación del ADN
- Baja concentración de ADN
- Alta concentración de sales
- Diseño erróneo de iniciadores o “primers“
Aplicaciones
- Técnicas biotecnológicas
- Secuenciación
- Filogenia
- Ancestría genética
- Genotipificación
- Ciencias Forenses
Temas relacionados
Cómo citar: Checa Rojas, A. (2016, 22 de Junio ) PCR: Reacción en cadena de la polimerasa. Conogasi, Conocimiento para la vida. Fecha de consulta: Abril 19, 2024
Esta obra está disponible bajo una licencia de Creative Commons Reconocimiento-No Comercial Compartir Igual 4.0





Deja un comentario
2 Comentarios en "PCR: Reacción en cadena de la polimerasa"
[…] Reactivos […]
hola